Estructura terciaria de proteínas - Protein tertiary structure

La estructura terciaria de la proteína es la forma tridimensional de una proteína . La estructura terciaria tendrá un único "esqueleto" de cadena polipeptídica con una o más estructuras secundarias proteicas , los dominios proteicos . Las cadenas laterales de aminoácidos pueden interactuar y unirse de varias formas. Las interacciones y los enlaces de las cadenas laterales dentro de una proteína particular determinan su estructura terciaria. La estructura terciaria de la proteína se define por sus coordenadas atómicas . Estas coordenadas pueden referirse a un dominio de proteína o a la estructura terciaria completa. Varias estructuras terciarias pueden plegarse en una estructura cuaternaria .
Historia
La ciencia de la estructura terciaria de las proteínas ha pasado de una hipótesis a una de definición detallada. Aunque Emil Fischer había sugerido que las proteínas estaban formadas por cadenas polipeptídicas y cadenas laterales de aminoácidos, fue Dorothy Maud Wrinch quien incorporó la geometría en la predicción de las estructuras de las proteínas . Wrinch demostró esto con el modelo Cyclol , la primera predicción de la estructura de una proteína globular . Los métodos contemporáneos son capaces de determinar, sin predicción, estructuras terciarias con un margen de 5 Å (0,5 nm) para proteínas pequeñas (<120 residuos) y, en condiciones favorables, predicciones confiables de estructuras secundarias .
Determinantes
Estabilidad de los estados nativos
Termoestabilidad
Una proteína plegada en su estado nativo o conformación nativa típicamente tiene una energía libre de Gibbs más baja (una combinación de entalpía y entropía ) que la conformación desplegada. Una proteína tenderá hacia conformaciones de baja energía, lo que determinará el pliegue de la proteína en el entorno celular . Debido a que muchas conformaciones similares tendrán energías similares, las estructuras de las proteínas son dinámicas y fluctúan entre una gran cantidad de estas estructuras similares.
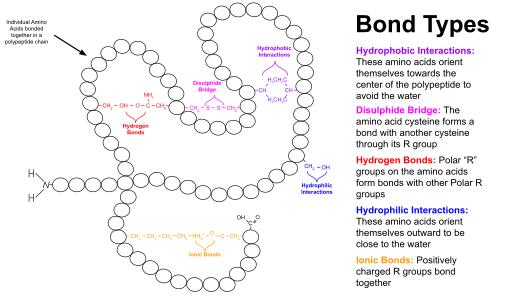
Las proteínas globulares tienen un núcleo de residuos de aminoácidos hidrófobos y una región superficial de residuos hidrófilos cargados, expuestos al agua . Esta disposición puede estabilizar las interacciones dentro de la estructura terciaria. Por ejemplo, en las proteínas secretadas , que no están bañadas en el citoplasma , los enlaces disulfuro entre los residuos de cisteína ayudan a mantener la estructura terciaria. Existe una similitud de estructuras terciarias estables observadas en proteínas de función diversa y evolución diversa . Por ejemplo, el cilindro TIM , llamado así por la enzima triosafosfatoisomerasa , es una estructura terciaria común, al igual que la estructura en espiral , dimérica , altamente estable . Por tanto, las proteínas pueden clasificarse según las estructuras que contienen. Las bases de datos de proteínas que utilizan tal clasificación incluyen SCOP y CATH .
Trampas cinéticas
La cinética de plegado puede atrapar una proteína en una conformación de alta energía , es decir, una conformación intermedia de alta energía bloquea el acceso a la conformación de menor energía. La conformación de alta energía puede contribuir a la función de la proteína. Por ejemplo, la proteína hemaglutinina de la influenza es una única cadena polipeptídica que, cuando se activa, se escinde proteolíticamente para formar dos cadenas polipeptídicas. Las dos cadenas se mantienen en una conformación de alta energía. Cuando el pH local desciende, la proteína experimenta un reordenamiento conformacional energéticamente favorable que le permite penetrar la membrana de la célula huésped .
Metaestabilidad
Algunas estructuras de proteínas terciarias pueden existir en estados de larga duración que no son el estado más estable esperado. Por ejemplo, muchas serpinas (inhibidores de serina proteasa) muestran esta metaestabilidad . Sufren un cambio conformacional cuando una proteasa corta un bucle de la proteína .
Proteínas chaperonas
Se asume comúnmente que el estado nativo de una proteína es también el más termodinámicamente estable y que una proteína alcanzará su estado nativo, dada su cinética química , antes de ser traducida . Las chaperonas proteicas dentro del citoplasma de una célula ayudan a un polipéptido recién sintetizado a alcanzar su estado nativo. Algunas proteínas chaperonas son muy específicas en su función, por ejemplo, la proteína disulfuro isomerasa ; otros son generales en su función y pueden ayudar a la mayoría de las proteínas globulares, por ejemplo, el sistema de proteínas procariotas GroEL / GroES y las proteínas eucariotas homólogas de choque térmico (el sistema Hsp60 / Hsp10).
Entorno citoplasmático
La predicción de la estructura terciaria de la proteína se basa en conocer la estructura primaria de la proteína y comparar la posible estructura terciaria predicha con estructuras terciarias conocidas en los bancos de datos de proteínas . Esto solo tiene en cuenta el entorno citoplasmático presente en el momento de la síntesis de proteínas en la medida en que un entorno citoplasmático similar también puede haber influido en la estructura de las proteínas registradas en el banco de datos de proteínas.
La Unión del ligando
La estructura de una proteína, por ejemplo, una enzima , puede cambiar tras la unión de sus ligandos naturales, por ejemplo, un cofactor . En este caso, la estructura de la proteína unida al ligando se conoce como holoestructura, de la proteína no unida como apoestructura.
Estructura estabilizada por la formación de enlaces débiles entre las cadenas laterales de aminoácidos - Determinada por el plegamiento de la cadena polipeptídica sobre sí misma (los residuos no polares se encuentran dentro de la proteína, mientras que los residuos polares se encuentran principalmente en el exterior) - La envoltura de la proteína acerca a la proteína y se relaciona a-to ubicado en regiones distantes de la secuencia - La adquisición de la estructura terciaria conduce a la formación de bolsas y sitios adecuados para el reconocimiento y la unión de moléculas específicas (bioespecificidad)
Determinación
El conocimiento de la estructura terciaria de las proteínas globulares solubles es más avanzado que el de las proteínas de membrana porque las primeras son más fáciles de estudiar con la tecnología disponible.
Cristalografía de rayos X
La cristalografía de rayos X es la herramienta más común utilizada para determinar la estructura de las proteínas . Proporciona una alta resolución de la estructura pero no da información sobre la flexibilidad conformacional de la proteína .
RMN
La proteína NMR da una resolución comparativamente más baja de la estructura de la proteína. Se limita a proteínas más pequeñas. Sin embargo, puede proporcionar información sobre cambios conformacionales de una proteína en solución.
Microscopía electrónica criogénica
La microscopía electrónica criogénica (crio-EM) puede proporcionar información sobre la estructura terciaria y cuaternaria de una proteína. Es particularmente adecuado para proteínas grandes y complejos simétricos de subunidades de proteínas .
Interferometría de polarización dual
La interferometría de polarización dual proporciona información complementaria sobre las proteínas capturadas en la superficie. Ayuda a determinar los cambios de estructura y conformación a lo largo del tiempo.
Proyectos
Algoritmo de predicción
El proyecto Folding @ home de la Universidad de Stanford es un esfuerzo de investigación de computación distribuida que utiliza aproximadamente 5 petaFLOPS (≈10 x86 petaFLOPS) de computación disponible. Su objetivo es encontrar un algoritmo que predecirá consistentemente las estructuras terciarias y cuaternarias de las proteínas, dada la secuencia de aminoácidos de la proteína y sus condiciones celulares.
Se puede encontrar una lista de software para la predicción de la estructura terciaria de proteínas en Lista de software de predicción de la estructura de proteínas .
Enfermedades por agregación de proteínas
La agregación de proteínas enfermedades tales como la enfermedad de Alzheimer y la enfermedad de Huntington y priones enfermedades como la encefalopatía espongiforme bovina pueden entenderse mejor mediante la construcción (y reconstruir) modelos de enfermedad . Esto se hace causando la enfermedad en animales de laboratorio, por ejemplo, administrando una toxina , como MPTP, para causar la enfermedad de Parkinson, o mediante manipulación genética . La predicción de la estructura de las proteínas es una nueva forma de crear modelos de enfermedades, que pueden evitar el uso de animales.
Proyecto de recuperación de estructuras terciarias de proteínas (CoMOGrad)
Hacer coincidir los patrones en la estructura terciaria de una proteína dada con una gran cantidad de estructuras terciarias de proteínas conocidas y recuperar las más similares en orden de clasificación está en el corazón de muchas áreas de investigación, como la predicción de funciones de proteínas nuevas, el estudio de la evolución, el diagnóstico de enfermedades, el descubrimiento de fármacos, diseño de anticuerpos, etc. El proyecto CoMOGrad en BUET es un esfuerzo de investigación para diseñar un método extremadamente rápido y muy preciso para la recuperación de la estructura terciaria de proteínas y desarrollar una herramienta en línea basada en los resultados de la investigación.
Ver también
Referencias
enlaces externos
- Banco de datos de proteínas
- Visualización, análisis y superposición de estructuras de proteínas en 3D.
- Alfabeto de estructuras proteicas.
- Visualización, análisis y superposición de estructuras de proteínas en 3D.
- Curso basado en WWW que enseña bioinformática proteica elemental
- Evaluación crítica de la predicción de estructuras (CASP)
- Clasificación estructural de proteínas (SCOP)
- Clasificación de la estructura de la proteína CATH
- Software DALI / FSSP y base de datos de estructuras proteicas superpuestas
- Núcleos estructurales invariantes TOPOFIT-DB entre proteínas
- PDBWiki - Página de inicio de PDBWiki - un sitio web para la anotación comunitaria de estructuras PDB.