Mapa de contacto de proteínas - Protein contact map
Un mapa de contacto de proteínas representa la distancia entre todos los posibles pares de residuos de aminoácidos de una estructura de proteína tridimensional utilizando una matriz bidimensional binaria . Para dos residuos y , el elemento de la matriz es 1 si los dos residuos están más cerca de un umbral predeterminado y 0 en caso contrario. Se han propuesto varias definiciones de contacto: la distancia entre el átomo C α- C α con umbral 6-12 Å ; distancia entre átomos de C β- C β con umbral de 6-12 Å (C α se usa para glicina ); y distancia entre los centros de masa de la cadena lateral .
Visión general
Los mapas de contacto proporcionan una representación más reducida de la estructura de una proteína que sus coordenadas atómicas 3D completas. La ventaja es que los mapas de contactos son invariables a las rotaciones y traducciones. Son más fáciles de predecir mediante métodos de aprendizaje automático . También se ha demostrado que en determinadas circunstancias (por ejemplo, bajo contenido de contactos predichos erróneamente) es posible reconstruir las coordenadas 3D de una proteína utilizando su mapa de contacto.
Los mapas de contacto también se utilizan para la superposición de proteínas y para describir similitudes entre estructuras de proteínas. Se predicen a partir de la secuencia de proteínas o se calculan a partir de una estructura determinada.
Predicción del mapa de contacto
Con la disponibilidad de un gran número de secuencias genómicas, resulta factible analizar dichas secuencias en busca de residuos coevolucionantes . La eficacia de este enfoque se debe al hecho de que es más probable que una mutación en la posición i de una proteína se asocie con una mutación en la posición j que con una mutación inversa en i si ambas posiciones están funcionalmente acopladas (por ejemplo, al participar en un dominio enzimático, o por ser adyacente en una proteína plegada, o incluso por ser adyacente en un oligómero de esa proteína).
Existen varios métodos estadísticos para extraer de un alineamiento de secuencia múltiple tales pares de residuos acoplados: frecuencias observadas versus esperadas de pares de residuos (OMES); la correlación de sustitución basada en McLachlan (McBASC); análisis de acoplamiento estadístico ; Métodos basados en información mutua (MI); y recientemente análisis de acoplamiento directo (DCA).
Los algoritmos de aprendizaje automático han podido mejorar los métodos de análisis de MSA, especialmente para proteínas no homólogas (es decir, MSA superficiales).
Los mapas de contacto predichos se han utilizado en la predicción de proteínas de membrana donde se dirigen las interacciones hélice-hélice.
Parcela HB
El conocimiento de la relación entre la estructura de una proteína y su comportamiento dinámico es esencial para comprender la función de la proteína. La descripción de la estructura tridimensional de una proteína como una red de interacciones de enlace de hidrógeno ( gráfico HB ) se introdujo como una herramienta para explorar la estructura y función de la proteína. Al analizar la red de interacciones terciarias, se puede investigar la posible propagación de información dentro de una proteína.
El gráfico HB ofrece una forma sencilla de analizar la estructura secundaria y la estructura terciaria de las proteínas . Los enlaces de hidrógeno que estabilizan los elementos estructurales secundarios ( enlaces de hidrógeno secundarios ) y los formados entre residuos de aminoácidos distantes , definidos como enlaces de hidrógeno terciarios , se pueden distinguir fácilmente en la gráfica HB, por lo que se pueden identificar los residuos de aminoácidos implicados en la estabilización de la estructura y función de las proteínas .
Características
El gráfico distingue entre las interacciones de enlace de hidrógeno cadena principal-cadena principal, cadena principal-cadena lateral y cadena lateral-cadena lateral . Enlaces de hidrógeno bifurcados y enlaces de hidrógeno múltiples entre residuos de aminoácidos; y los enlaces de hidrógeno intra e intercadena también se indican en los gráficos. Tres clases de enlaces de hidrógeno se distinguen por codificación de colores; enlaces de hidrógeno cortos (distancia menor de 2.5 Å entre donante y aceptor), intermedios (entre 2.5 Å y 3.2 Å) y largos (mayor de 3.2 Å).
Elementos de estructura secundaria en la parcela HB
En las representaciones de la gráfica HB, los patrones característicos de los elementos de la estructura secundaria se pueden reconocer fácilmente, como sigue:
- Las hélices se pueden identificar como tiras directamente adyacentes a la diagonal.
- Las láminas beta antiparalelas aparecen en la gráfica HB como diagonales cruzadas.
- Las hojas beta paralelas aparecen en el gráfico HB como paralelas a la diagonal.
- Los bucles aparecen como roturas en la diagonal entre los motivos de la hoja beta en diagonal cruzada .
Ejemplos de uso
Citocromo P450s
Los P450s citocromo (P450s) son xenobióticos -metabolizing membrana -bound heme que contiene enzimas que el uso molecular de oxígeno y electrones de NADPH citocromo P450 reductasa para oxidar sus sustratos . CYP2B4 , un miembro de la familia del citocromo P450, es la única proteína dentro de esta familia, cuya estructura de rayos X tanto en forma abierta 11 como cerrada 12 está publicada. La comparación de las estructuras abiertas y cerradas de las estructuras de CYP2B4 revela un reordenamiento conformacional a gran escala entre los dos estados, con el mayor cambio conformacional alrededor de los residuos 215-225, que está ampliamente abierto en estado libre de ligando y cerrado después de la unión del ligando; y la región alrededor del bucle C cerca del hemo.
El examen de la gráfica de HB del estado cerrado y abierto de CYP2B4 reveló que el reordenamiento de los enlaces de hidrógeno terciarios estaba en excelente acuerdo con el conocimiento actual del ciclo catalítico del citocromo P450 .
El primer paso en el ciclo catalítico de P450 se identifica como unión al sustrato. La unión preliminar de un ligando cerca de la entrada rompe los enlaces de hidrógeno S212-E474, S207-H172 en la forma abierta de CYP2B4 y se forman enlaces de hidrógeno E218-A102, Q215-L51 que fijan la entrada en la forma cerrada como revela el gráfico de HB .
El segundo paso es la transferencia del primer electrón de NADPH a través de una cadena de transferencia de electrones. Para la transferencia de electrones se produce un cambio conformacional que desencadena la interacción del P450 con el citocromo P450 reductasa NADPH. Romper los enlaces de hidrógeno entre S128-N287, S128-T291, L124-N287 y formar S96-R434, A116-R434, R125-I435, D82-R400 en el sitio de unión de la reductasa del citocromo P450 de NADPH , como se ve en el gráfico de HB, transformar CYP2B4 a un estado de conformación, donde se produce la unión de la reductasa del citocromo P450 NADPH.
En el tercer paso, el oxígeno ingresa a CYP2B4 en el estado cerrado, el estado en el que los enlaces de hidrógeno recién formados S176-T300, H172-S304, N167-R308 abren un túnel que tiene exactamente el tamaño y la forma de una molécula de oxígeno .
Familia lipocalina
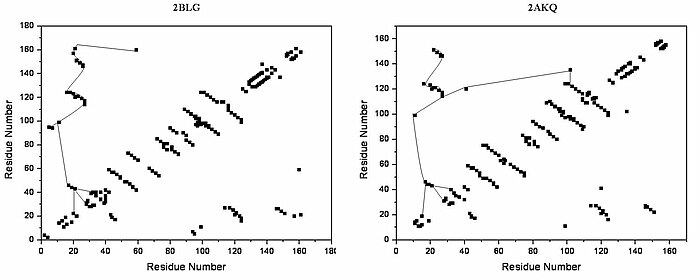
La familia de las lipocalinas es una familia grande y diversa de proteínas con funciones como pequeñas moléculas transportadoras hidrófobas . La beta-lactoglobulina es un miembro típico de la familia de las lipocalinas. Se encontró que la beta-lactoglobulina tiene un papel en el transporte de ligandos hidrófobos como el retinol o los ácidos grasos . Su estructura cristalina se determinó [por ejemplo, Qin, 1998] con diferentes ligandos y también en forma libre de ligando. Las estructuras cristalinas determinadas hasta ahora revelan que la lipocalina típica contiene un barril antiparalelo de ocho hebras dispuesto para formar una cavidad central cónica en la que se une el ligando hidrófobo. La estructura de la beta-lactoglobulina revela que la estructura en forma de barril con la cavidad central de la proteína tiene una "entrada" rodeada por cinco bucles beta con centros alrededor de 26, 35, 63, 87 y 111, que experimentan un cambio conformacional. durante la unión del ligando y cerrar la cavidad.
La forma general de la beta-lactoglobulina es característica de la familia de las lipocalinas. En ausencia de hélices alfa , la diagonal principal casi desaparece y las diagonales cruzadas que representan las hojas beta dominan la trama. En la gráfica se puede encontrar un número relativamente bajo de enlaces de hidrógeno terciarios, con tres regiones de alta densidad, una de las cuales está conectada a un bucle en los residuos alrededor de 63, un segundo está conectado al bucle alrededor de 87 y una tercera región que está conectado a las regiones 26 y 35. El quinto bucle alrededor de 111 está representado solo un enlace de hidrógeno terciario en el gráfico HB.
En la estructura tridimensional, los enlaces de hidrógeno terciarios se forman (1) cerca de la entrada, directamente involucrados en el reordenamiento conformacional durante la unión del ligando; y (2) en la parte inferior del "barril". Los gráficos de HB de las formas abierta y cerrada de beta-lactoglobulina son muy similares, todos los motivos únicos pueden reconocerse en ambas formas. La diferencia en los gráficos de HB de forma abierta y unida a ligando muestra pocos cambios individuales importantes en el patrón de enlace de hidrógeno terciario. Especialmente, la formación de enlaces de hidrógeno entre Y20-E157 y S21-H161 en forma cerrada podría ser crucial en el reordenamiento conformacional. Estos enlaces de hidrógeno se encuentran en la parte inferior de la cavidad, lo que sugiere que el cierre de la entrada de una lipocalina comienza cuando un ligando alcanzó el fondo de la cavidad y rompió los enlaces de hidrógeno R123-Y99, R123-T18 y V41-Q120. Se sabe que las lipocalinas tienen una similitud de secuencia muy baja con una gran similitud estructural. Las únicas regiones conservadas son exactamente la región alrededor de 20 y 160 con un papel desconocido.
Ver también
- Parcela de Ramachandran
- Clasificación estructural de proteínas
- CATH
- Parcela HB
- Gráfico de puntos (bioinformática)
- Matriz de auto-semejanza
Referencias
enlaces externos
- DESTILADO : predicción de características estructurales de proteínas (incluidos mapas de contacto de residuos de proteínas)
- Herramientas de proteómica estructural : incluye mapas de contacto de aminoácidos
- ProfCon - predicción de contactos entre residuos
- TMHcon : predicción de contactos hélice-hélice específicamente dentro de las partes transmembrana de las proteínas de membrana
- TMhit : un nuevo método de predicción de la interacción hélice-hélice transmembrana basado en contactos de residuos
- CMAPpro : un servidor de predicción de mapas de contacto de proteínas
- [1] —Una herramienta para la visualización de mapas de contacto de proteínas en jerseysforcheapshop