Acoplamiento dipolar residual - Residual dipolar coupling
El acoplamiento dipolar residual entre dos espines en una molécula ocurre si las moléculas en solución exhiben un alineamiento parcial que conduce a un promedio incompleto de acoplamientos dipolares espacialmente anisotrópicos .
La alineación molecular parcial conduce a un promedio incompleto de interacciones magnéticas anisotrópicas, como la interacción dipolo-dipolo magnético (también llamada acoplamiento dipolar), la anisotropía de desplazamiento químico o la interacción cuadripolo eléctrica . Las llamadas interacciones magnéticas anisotrópicas residuales resultantes son cada vez más importantes en la espectroscopia de RMN biomolecular .
Historia y obras pioneras
La espectroscopía de RMN en medios parcialmente orientados se descubrió por primera vez en 1963, y en un artículo muy fundamental, Alfred Saupe también pudo presentar la teoría esencial para describir y comprender los fenómenos observables solo un año después. Después de esta iniciación, se informó de una inundación de espectros de RMN en varias fases cristalinas líquidas (véase, por ejemplo ).
Una segunda técnica para la alineación parcial que no está limitada por una anisotropía mínima es la alineación inducida por deformación en un gel (SAG), basada en el trabajo pionero de Deloche y Samulski. La técnica se utilizó ampliamente para estudiar las propiedades de los geles poliméricos por medio de RMN de deuterio de alta resolución, pero solo recientemente se utilizó la alineación del gel para inducir RDC en moléculas disueltas en el gel. SAG permite el escalado de alineación sin restricciones en un amplio rango y se puede utilizar para disolventes acuosos y orgánicos, según el polímero utilizado. Como un primer ejemplo en disolventes orgánicos, las mediciones de RDC en poliestireno estirado (PS) geles hinchados en CDCl 3 se informaron como un método de alineación prometedor.
En 1995, James H. Prestegard y colaboradores demostraron que los espectros de RMN de ciertas proteínas (en este caso, la cianomemioglobina, que tiene una susceptibilidad paramagnética muy anisotrópica ), tomados a un campo muy alto, pueden contener datos que pueden complementar de manera útil los NOE para determinar un nivel terciario. pliegue.
En 1996 y 1997, Adriaan Bax y colaboradores midieron las CDR en una proteína diamagnética ( ubiquitina ). Los resultados estuvieron de acuerdo con las estructuras cristalinas.
Física
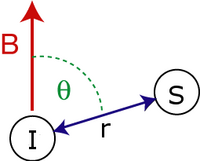
El acoplamiento dipolar secular hamiltoniano de dos espines , y está dado por:
dónde
- es la constante de Planck reducida .
- y son las relaciones giromagnéticas de espín y espín, respectivamente.
- es la distancia entre espines.
- es el ángulo entre el vector entre espines y el campo magnético externo .
- y son vectores de operadores de espín .
La ecuación anterior se puede reescribir de la siguiente forma:
dónde
En solución isotrópica, el volteo molecular reduce el valor medio de a cero. Por tanto, no observamos ningún acoplamiento dipolar. Si la solución no es isotrópica, entonces el valor promedio de puede ser diferente de cero y se pueden observar acoplamientos residuales .
Tenga en cuenta que este acoplamiento dipolar residual puede ser positivo o negativo, dependiendo del rango de ángulos muestreados.
Además de la distancia estática y la información angular, los RDC pueden contener información sobre el movimiento interno de una molécula. A cada átomo de una molécula se le puede asociar un tensor de movimiento B , que se puede calcular a partir de las RDC de acuerdo con la siguiente relación:
donde A es el tensor de alineación molecular . Las filas de B contienen los tensores de movimiento para cada átomo. Los tensores de movimiento también tienen cinco grados de libertad . A partir de cada tensor de movimiento, se pueden calcular 5 parámetros de interés. Las variables S i 2 , η i , α i , β i y γ i se utilizan para denotar estos 5 parámetros para el átomo i. S i 2 es la magnitud del átomo de movimiento i de; η i es una medida de la anisotropía del movimiento del átomo i; α i y β i están relacionados con las coordenadas polares del vector de enlace expresado en el marco de referencia arbitrario inicial (es decir, el marco PDB). Si el movimiento del átomo es anisotrópico (es decir, η i = 0), el parámetro final, γ i mide la orientación principal del movimiento.
Tenga en cuenta que los parámetros de movimiento derivados de RDC son medidas locales.
Medición
Cualquier medición de RDC en solución consta de dos pasos, alineación de moléculas y estudios de RMN:
Métodos para alinear moléculas.
Para las moléculas diamagnéticas con intensidades de campo moderadas, las moléculas tienen poca preferencia en la orientación, las muestras de volteo tienen una distribución casi isotrópica y los acoplamientos dipolares promedio llegan a cero. En realidad, la mayoría de las moléculas tienen orientaciones preferidas en presencia de un campo magnético, porque la mayoría tienen tensores de susceptibilidad magnética anisotrópicos , Χ.
El método es más adecuado para sistemas con valores altos de tensor de susceptibilidad magnética. Esto incluye: complejo proteína-ácido nucleico, ácidos nucleicos , proteínas con gran número de residuos aromáticos , proteínas que contienen porfirina y proteínas de unión a metales (el metal puede ser reemplazado por lantánidos ).
Para una molécula totalmente orientada, el acoplamiento dipolar para un grupo amida 1 H- 15 N sería superior a 20 kHz , y un par de protones separados por 5 Å tendrían un acoplamiento de hasta ~ 1 kHz. Sin embargo, el grado de alineación logrado mediante la aplicación de un campo magnético es tan bajo que los acoplamientos dipolares 1 H- 15 N o 1 H- 13 C más grandes son <5 Hz. Por lo tanto, se han diseñado muchos medios de alineación diferentes:
- Bicelas lipídicas (con gran susceptibilidad magnética): las RDC medidas fueron del orden de cientos de Hz.
- Bicelas cristalinas líquidas: las RDC medidas estaban entre -40 y +20 Hz.
- Virus en forma de bastón, incluido el bacteriófago filamentoso (gran susceptibilidad magnética anisotrópica).
- Nanotubos de ADN (compatibles con detergentes utilizados para solubilizar proteínas de membrana)
Experimentos de RMN
Existen numerosos métodos que se han diseñado para medir con precisión la constante de acoplamiento entre núcleos. Se han clasificado en dos grupos: métodos basados en la frecuencia donde la separación de los centros de los picos (división) se mide en un dominio de frecuencia, y métodos basados en la intensidad donde el acoplamiento se extrae de la intensidad de resonancia en lugar de dividirse. Los dos métodos se complementan, ya que cada uno de ellos está sujeto a un tipo diferente de errores sistemáticos. Aquí están los ejemplos prototípicos de experimentos de RMN que pertenecen a cada uno de los dos grupos:
- Métodos de intensidad : experimento cuantitativo de modulación J y métodos de modulación de fase
- métodos de resolución de frecuencia : SCE- HSQC , E. COSY y experimentos selectivos de estado de giro
Biología estructural
La medición de RDC proporciona información sobre el plegamiento global de la proteína o del complejo proteico. A diferencia de las determinaciones tradicionales de estructura de RMN basadas en NOE , los RDC proporcionan información estructural a larga distancia. También proporciona información sobre la dinámica de las moléculas en escalas de tiempo más lentas que los nanosegundos.
Estudios de estructura biomolecular
La mayoría de los estudios de RMN de la estructura de la proteína se basan en el análisis del efecto Nuclear Overhauser , NOE, entre diferentes protones de la proteína. Debido a que el NOE depende de la sexta potencia invertida de la distancia entre los núcleos, r −6 , los NOE se pueden convertir en restricciones de distancia que se pueden usar en cálculos de estructura de tipo de dinámica molecular . Los RDC proporcionan restricciones de orientación en lugar de restricciones de distancia, y tienen varias ventajas sobre los NOE:
- Los RDC brindan información sobre el ángulo relativo al campo magnético externo, lo que significa que pueden brindar información sobre la orientación relativa de las partes de la molécula que están muy separadas en la estructura.
- En moléculas grandes (> 25 kDa), a menudo es difícil registrar NOE debido a la difusión de espín . Esto no es un problema con los RDC.
- El análisis de un gran número de NOE puede llevar mucho tiempo.
Siempre que se disponga de un conjunto muy completo de RDC, se ha demostrado para varios sistemas modelo que las estructuras moleculares pueden calcularse exclusivamente basándose en estas interacciones anisotrópicas, sin recurrir a restricciones NOE. Sin embargo, en la práctica, esto no es alcanzable y RDC se utiliza principalmente para perfeccionar una estructura determinada por los datos de NOE y J-acoplamiento . Un problema con el uso de acoplamientos dipolares en la determinación de la estructura es que un acoplamiento dipolar no describe de forma única una orientación de vector internuclear. Además, si se dispone de un conjunto muy pequeño de acoplamientos dipolares, el refinamiento puede conducir a una estructura peor que la original. Para una proteína con N aminoácidos, la restricción de 2N RDC para la columna vertebral es el mínimo necesario para un refinamiento preciso.
El contenido de información de una medición de RDC individual para un vector de enlace específico (como un enlace NH de cadena principal específico en una molécula de proteína) puede entenderse mostrando la curva objetivo que traza direcciones de concordancia perfecta entre el valor de RDC observado y el valor calculado del modelo. Tal curva (ver figura) tiene dos ramas simétricas que se encuentran en una esfera con su eje polar a lo largo de la dirección del campo magnético. Su altura desde el ecuador de la esfera depende de la magnitud del valor RDC y su forma depende de la "rombicidad" (asimetría) del tensor de alineación molecular. Si la alineación molecular fuera completamente simétrica alrededor de la dirección del campo magnético, la curva objetivo consistiría simplemente en dos círculos en el mismo ángulo de los polos que el ángulo que forma el vector de enlace específico con el campo magnético aplicado.
En el caso de moléculas alargadas como el ARN , donde la información de torsión local y las distancias cortas no son suficientes para restringir las estructuras, las mediciones de RDC pueden proporcionar información sobre las orientaciones de enlaces químicos específicos a lo largo de un ácido nucleico con respecto a un solo marco de coordenadas. Particularmente, las moléculas de ARN son pobres en protones y la superposición de resonancias de ribosa dificulta mucho el uso de datos de acoplamiento J y NOE para determinar la estructura. Además, se pueden detectar RDC entre núcleos con una distancia superior a 5-6 Å. Esta distancia es demasiado para la generación de señal NOE. Esto se debe a que RDC es proporcional a r −3, mientras que NOE es proporcional a r −6 .
También se ha demostrado que las mediciones de RDC son extremadamente útiles para una determinación rápida de las orientaciones relativas de unidades de estructuras conocidas en proteínas. En principio, la orientación de una subunidad estructural, que puede ser tan pequeña como un giro de una hélice o tan grande como un dominio completo, puede establecerse a partir de tan solo cinco RDC por subunidad.
Dinámica proteica
Como un RDC proporciona información promediada espacial y temporalmente sobre un ángulo entre el campo magnético externo y un vector de enlace en una molécula, puede proporcionar información geométrica rica sobre la dinámica lenta (> 10-9 s) en las proteínas. En particular, debido a su dependencia radial, la RDC es particularmente sensible a los procesos angulares de gran amplitud. Un ejemplo temprano de Tolman et al. encontraron estructuras de mioglobina previamente publicadas insuficientes para explicar los datos de RDC medidos, y diseñaron un modelo simple de dinámica lenta para remediar esto. Sin embargo, para muchas clases de proteínas, incluidas las proteínas intrínsecamente desordenadas , el análisis de las RDC se vuelve más complicado, ya que definir un marco de alineación no es trivial. Dos artículos recientes describen un remedio prometedor para el problema, al eludir la necesidad de definir explícitamente el marco de alineación.
Ver también
- Interacción dipolo-dipolo magnético
- Anisotropía de desplazamiento químico residual (rCSA)
- Resonancia magnética nuclear de estado sólido (ssNMR)
Referencias
Otras lecturas
Libros :
- Emsley, JW; Lindon, JC NMR Spectroscopy usando disolventes de cristal líquido; Pergamon Press: Oxford, Reino Unido, 1975.
Artículos de revisión :
- Ad Bax y Alexander Grishaev, Current Opinion in Structural Biology , 15: 563–570 (2005)
- Rebecca S. Lipsitz y Nico Tjandra, Annu. Rev. Biophys. Biomol. Struct . 33: 387–413 (2004)
Papeles clásicos :
- Saupe, A .; Englert, G. (1963). "Espectros de resonancia magnética nuclear de alta resolución de moléculas orientadas". Cartas de revisión física . 11 (10): 462–464. Código Bibliográfico : 1963PhRvL..11..462S . doi : 10.1103 / physrevlett.11.462 .
- Saupe, A (1964). "Kernresonanzen in kristallinen Flüssigkeiten und in kristallinflüssigen Lösungen. Teil I". Zeitschrift für Naturforschung . 19a (2): 161-171. Código bibliográfico : 1964ZNatA..19..161S . doi : 10.1515 / zna-1964-0201 . S2CID 94943333 .
- Deloche, B .; Samulski, ET (1981). "Orden de orientación similar a nemático de corto alcance en elastómeros tensados: un estudio de resonancia magnética de deuterio". Macromoléculas . 14 (3): 575–581. Código bibliográfico : 1981MaMol..14..575D . doi : 10.1021 / ma50004a024 .
- Tjandra, Nico; Bax, Ad (1997). "Medición directa de distancias y ángulos en biomoléculas por RMN en medio líquido cristalino diluido". Ciencia . 278 (5340): 1111–1114. Bibcode : 1997Sci ... 278.1111T . doi : 10.1126 / science.278.5340.1111 . PMID 9353189 .
- Tjandra, N .; Omichinski, JG; Gronenborn, AM; Clore, GM; Bax, A. (1997). "Uso de acoplamientos dipolares 1H-15N y 1H-13C en la determinación de la estructura de macromoléculas orientadas magnéticamente en solución". Biología estructural de la naturaleza . 4 (9): 732–738. doi : 10.1038 / nsb0997-732 . PMID 9303001 . S2CID 22357937 .
- Tjandra, N .; Bax, A. (1997). "Medición de las contribuciones dipolares a las escisiones de 1JCH de la dependencia del campo magnético de la modulación J en espectros bidimensionales de RMN". J. Magn. Reson . 124 (2): 512–515. Código Bibliográfico : 1997JMagR.124..512T . doi : 10.1006 / jmre.1996.1088 . PMID 9169226 .
- Tolman, JR; et al. (1997). "Evidencia de RMN para movimientos colectivos lentos en cianomemioglobina". Biología estructural de la naturaleza . 4 (4): 292-297. doi : 10.1038 / nsb0497-292 . PMID 9095197 . S2CID 29605996 .
- Tolman, JR; Prestegard, JH (1996). "Un experimento de correlación cuantitativaJ para la medición precisa de acoplamientos One-Bond Amide15N – 1H en proteínas". J. Magn. Reson. B . 112 (3): 245–252. Código bibliográfico : 1996JMRB..112..245T . doi : 10.1006 / jmrb.1996.0138 . PMID 8921605 .
- Tolman, JR; Flanagan, JM; Kennedy, MA; Prestegard, JH (1995). "Interacciones de dipolos magnéticos nucleares en proteínas orientadas al campo: información para la determinación de la estructura en solución" . Proc. Natl. Acad. Sci. USA . 92 (20): 9279–9283. Código Bibliográfico : 1995PNAS ... 92.9279T . doi : 10.1073 / pnas.92.20.9279 . PMC 40968 . PMID 7568117 .
- Clore, GM (2000). "Acoplamiento preciso y rápido de complejos proteína-proteína sobre la base de datos de mejora de Overhauser nucleares intermoleculares y acoplamientos dipolares por minimización de cuerpo rígido" . Proc. Natl. Acad. Sci. USA . 97 (16): 9021–9025. Código Bibliográfico : 2000PNAS ... 97.9021C . doi : 10.1073 / pnas.97.16.9021 . PMC 16814 . PMID 10922057 .
- Sanders, CR; Liebre, BJ; Howard, KP; Prestegard, JH (1994). "Micelas de fosfolípidos orientadas magnéticamente como herramienta para el estudio de moléculas asociadas a la membrana". Prog. Nucl. Magn. Reson. Spectrosc . 26 : 421–444. doi : 10.1016 / 0079-6565 (94) 80012-x .
- Bastiaan, EW; Maclean, C .; Van Zijl, PCM; Bothner-By, AA (1987). "RMN de alta resolución de líquidos y gases: efectos de la alineación molecular inducida por campos magnéticos". Annu. Rep. NMR Spectrosc . Informes anuales sobre espectroscopia de RMN. 19 : 35–77. doi : 10.1016 / s0066-4103 (08) 60245-8 . ISBN 9780125053198.




![{\ Displaystyle H _ {\ mathrm {D}} = {\ frac {\ hbar ^ {2} \ gamma _ {I} \ gamma _ {S}} {4 \ pi r_ {IS} ^ {3}}} [ 1-3 \ cos ^ {2} \ theta] (3I_ {z} S_ {z} - {\ vec {I}} \ cdot {\ vec {S}})}](https://wikimedia.org/api/rest_v1/media/math/render/svg/64cb9a1de4ac67a4d920322838be224b4330f0b2)








![H _ {{\ mathrm {D}}} = D _ {{IS}} (\ theta) [2I_ {z} S_ {z} - (I_ {x} S_ {x} + I_ {y} S_ {y}) ] \!](https://wikimedia.org/api/rest_v1/media/math/render/svg/f1ef53aaf415fafa421f740336087b2fe905859d)
![{\ Displaystyle D_ {IS} (\ theta) = {\ frac {\ hbar ^ {2} \ gamma _ {I} \ gamma _ {S}} {4 \ pi r_ {IS} ^ {3}}} [ 1-3 \ cos ^ {2} \ theta]. \!}](https://wikimedia.org/api/rest_v1/media/math/render/svg/5304d624d47f35b40cf0fcb7e7cb645a5339c377)



