Oleandomicina - Oleandomycin
 | |
| Datos clinicos | |
|---|---|
| AHFS / Drugs.com | Nombres internacionales de medicamentos |
| Código ATC | |
| Identificadores | |
| |
| Número CAS | |
| PubChem CID | |
| ChemSpider | |
| UNII | |
| CHEMBL | |
| Número e | E704 (antibióticos) |
| Tablero CompTox ( EPA ) | |
| Tarjeta de información ECHA |
100.021.360 |
| Datos químicos y físicos | |
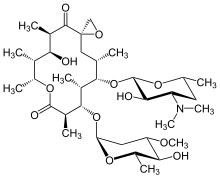
| Fórmula | C 35 H 61 N O 12 |
| Masa molar | 687,868 g · mol −1 |
| Modelo 3D ( JSmol ) | |
| |
| |
|
| |
La oleandomicina es un antibiótico macrólido . Se sintetiza a partir de cepas de Streptomyces antibioticus . Es más débil que la eritromicina .
Solía venderse bajo la marca Sigmamycine , combinado con tetraciclina , y fabricado por la empresa Rosa-Phytopharma en Francia .
Disponibilidad y uso médico
La oleandomicina se puede emplear para inhibir las actividades de las bacterias responsables de causar infecciones en el tracto respiratorio superior al igual que la eritromicina. Ambos pueden afectar a los géneros Staphylococcus y Enterococcus .
La CIM de oleandomicina es de 0,3-3 µg / ml para Staphylococcus aureus .
La oleandomicina está aprobada como antibiótico veterinario en algunos países. Ha sido aprobado como antibiótico para cerdos y aves de corral en los Estados Unidos. Sin embargo, actualmente solo está aprobado en los Estados Unidos para usos de producción.
Nombres de marca
- Mastalone - Oleandomicina, oxatetraciclina, neomicina - Zoetis Australia y Pfizer Animal Health
- Mastiguard - Oleandomicina, oxatetraciclina - Stockguard Animal Health
- Anteriormente vendido como Sigmamycine por Pfizer (Oleandomicina + Tetraciclina + Vitamina C )
Historia
Orígenes
La oleandomicina fue descubierta por primera vez como un producto de la bacteria Streptomyces antibioticus en 1954 por el Dr. Sobin, English y Celmer. En 1960, Hochstein logró determinar con éxito la estructura de la oleandomicina. Este macrólido se descubrió aproximadamente al mismo tiempo que sus parientes eritromicina y espiramicina.
Sigmamicina, fármaco combinado
El interés público en la oleandomicina alcanzó su punto máximo cuando Pfizer introdujo el fármaco combinado Sigmamycine en el mercado en 1956. Sigmamycine era un fármaco combinado de oleandomicina y tetraciclina respaldado por una importante campaña de marketing. De hecho, se afirmó que una mezcla 2: 1 de tetraciclina y oleandomicina tenía un efecto sinérgico sobre los estafilococos. También se afirmó que la mezcla sería eficaz en organismos que en su mayoría son resistentes a la tetraciclina u oleandomicina sola. Ambas afirmaciones fueron refutadas por hallazgos como los de Lawrence P. Garrod que no pudieron encontrar evidencia de que tales afirmaciones estuvieran debidamente fundamentadas. A principios de la década de 1970, los medicamentos combinados de Pfizer se retiraron del mercado.
Farmacología
Mecanismo de acción
La oleandomicina es un agente bacteriostático. Al igual que la eritromicina, la oleandomicina se une a la subunidad 50 de los ribosomas bacterianos, inhibiendo la finalización de proteínas vitales para la supervivencia y la replicación. Interfiere con la actividad de traducción, pero también con la formación de subunidades de los años 50.
Sin embargo, a diferencia de la eritromicina y sus eficaces derivados sintéticos, carece de un grupo 12-hidroxilo y un grupo 3-metoxi. Este cambio en la estructura puede afectar negativamente sus interacciones con las estructuras 50S y explicar por qué es un antibiótico menos potente.
Fuerza relativa
La oleandomicina es mucho menos eficaz que la eritromicina en las pruebas de concentración mínima inhibitoria bacteriana que involucran estafilococos o enterococos. Sin embargo, los antibióticos macrólidos pueden acumularse en órganos o células y este efecto puede prolongar la bioactividad de esta categoría de antibióticos incluso si su concentración en plasma está por debajo de lo que se considera capaz de un efecto terapéutico.
Química
Síntesis de policétidos
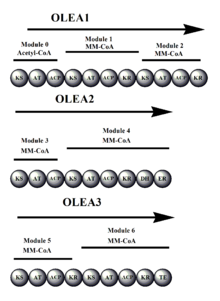
La oleandomicina sintasa (OLES) sigue la estructura del módulo de una sintasa de tipo I. La cadena de policétidos está unida a través de enlaces tioéster a los grupos SH de los dominios ACP y KS (no mostrados).
- El grupo de genes OLES1 codifica para los módulos 0-2, el módulo 0 que contiene una unidad de inicio de acetil-CoA y todos los restantes sin módulo llevan una unidad de elongación de metil malonil-CoA unida a su unidad de ceto sintasa.
- Códigos OLES2 para los módulos 3 y 4. El módulo 3 es notable por transportar potencialmente una cetoreductasa inactiva redox que es responsable de retener el carbonilo no reducido adyacente al carbono 8.
- Códigos OLES3 para módulos 5 y 6.
Las similitudes de la secuencia de aminoácidos entre OLES y la 6-desoxyeritronolida B sintasa (precursora de la eritromicina sintasa) muestran solo un 45% de identidad común. Tenga en cuenta que, a diferencia de la sintasa precursora de eritromicina, existe un KS en el dominio de carga de OLES.
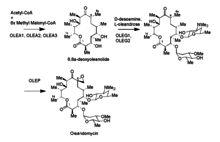
Sastrería post-PKS
Los genes OleG1 y G2 son responsables de las glicosiltransferasas que unen los azúcares característicos de la oleandomicina al macrólido. Estos azúcares se derivan de TDP-glucosa . OLEG1 transfiere dTDP-D-desoamina y OleG2 transfiere D-TDP-L-oleandrosa al anillo macrólido. La epoxidación que se produce después es de la enzima codificada por OleP, que podría ser homóloga con una enzima P450. El método por el cual se sospecha que los epoxidatos de OleP es una dihidroxilación seguida de la conversión de un grupo hidroxilo en un grupo fosfato que luego sale a través de un cierre de anillo nucleófilo por el otro grupo hidroxilo.