Tiorredoxina reductasa - Thioredoxin reductase
| Tiorredoxina reductasa | |
|---|---|
| Identificadores | |
| Símbolo | ? |
| InterPro | IPR005982 |
| PROSITE | PS00573 |
| SCOP2 | 1zof / SCOPe / SUPFAM |
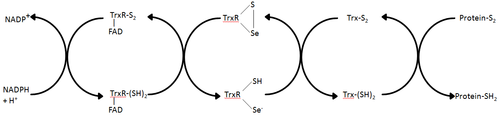
Las tiorredoxina reductasas ( TR , TrxR ) ( EC 1.8.1.9 ) son las únicas enzimas conocidas para reducir la tiorredoxina (Trx). Se han identificado dos clases de tiorredoxina reductasa: una clase en bacterias y algunas eucariotas y una en animales. En las bacterias, TrxR también cataliza la reducción de proteínas similares a la glutaredoxina conocidas como NrdH. Ambas clases son flavoproteínas que funcionan como homodímeros. Cada monómero contiene un grupo protésico FAD , un dominio de unión a NADPH y un sitio activo que contiene un enlace disulfuro activo redox .
Rol celular
La tiorredoxina reductasa es la única enzima que se sabe que cataliza la reducción de la tiorredoxina y, por tanto, es un componente central del sistema de las tiorredoxinas. Junto con la tiorredoxina (Trx) y NADPH, la descripción más general de este sistema es como un método para formar enlaces disulfuro reducidos en las células. Los electrones se toman de NADPH a través de TrxR y se transfieren al sitio activo de Trx, que continúa reduciendo los disulfuros de proteínas u otros sustratos. El sistema Trx existe en todas las células vivas y tiene una historia evolutiva ligada al ADN como material genético, defensa contra el daño oxidativo debido al metabolismo del oxígeno y señalización redox utilizando moléculas como el peróxido de hidrógeno y el óxido nítrico.
Diversidad
Dos clases de tiorredoxina reductasa han evolucionado de forma independiente:
- Se ha identificado un tipo de alto peso molecular (PM = ~ 55.000) que contiene un residuo de selenocisteína en su sitio activo en eucariotas superiores, incluidos los seres humanos. Este TxR está relacionado con la glutatión reductasa , tripanotiona reductasa , mercurio reductasa y lipoamida deshidrogenasa .
- Se ha identificado un tipo de bajo peso molecular (PM = ~ 35.000) en arqueas, bacterias y otros eucarios.
Estas dos clases de TrxR tienen solo ~ 20% de identidad de secuencia en la sección de secuencia primaria donde pueden alinearse de manera confiable. La reacción neta de ambas clases de TrxR es idéntica pero el mecanismo de acción de cada uno es distinto.
Los seres humanos expresan tres isoenzimas de tiorredoxina reductasa: tiorredoxina reductasa 1 (TrxR1, citosólica), tiorredoxina reductasa 2 (TrxR2, mitocondrial), tiorredoxina reductasa 3 (TrxR3, testicular específico). Cada isoenzima está codificada por un gen separado:
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Estructura
E. coli
En E. coli ThxR hay dos dominios de unión, uno para FAD y otro para NADPH . La conexión entre estos dos dominios es una hoja β antiparalela de dos hebras . Cada dominio individualmente es muy similar a los dominios análogos en la glutatión reductasa y la lipoamida deshidrogenasa, pero la orientación relativa de estos dominios en ThxR se rota 66 grados. Esto se vuelve significativo en el mecanismo de acción de la enzima que se describe a continuación. ThxR homo-dimeriza con la interfaz entre los dos monómeros formada por tres hélices alfa y dos bucles. Cada monómero puede unirse por separado a una molécula de tiorredoxina .
Mamífero
La estructura de TrxR de mamíferos es similar a la de E. coli . Contiene un dominio de unión de FAD y NADPH y una interfaz entre dos subunidades de monómero. En el ThxR de mamífero hay una inserción en el dominio de unión de FAD entre dos hélices alfa que forma un pequeño par de cadenas beta. El disulfuro activo de la enzima se encuentra en una de estas hélices y, por lo tanto, el enlace disulfuro activo está ubicado en el dominio FAD y no en el dominio NADPH como en E. coli y otros procariotas .
Mecanismo
E. coli
En E. coli ThxR, la orientación espacial de los dominios FAD y NADPH es tal que los anillos activos redox de FAD y NADPH no están muy próximos entre sí. Cuando el dominio FAD de E. coli se gira 66 grados con el dominio NADPH permaneciendo fijo, los dos grupos protésicos se mueven en estrecho contacto permitiendo que los electrones pasen de NADPH a FAD y luego al enlace disulfuro del sitio activo. Los residuos del sitio activo conservados en E. coli son -Cys-Ala-Thr-Cys-.
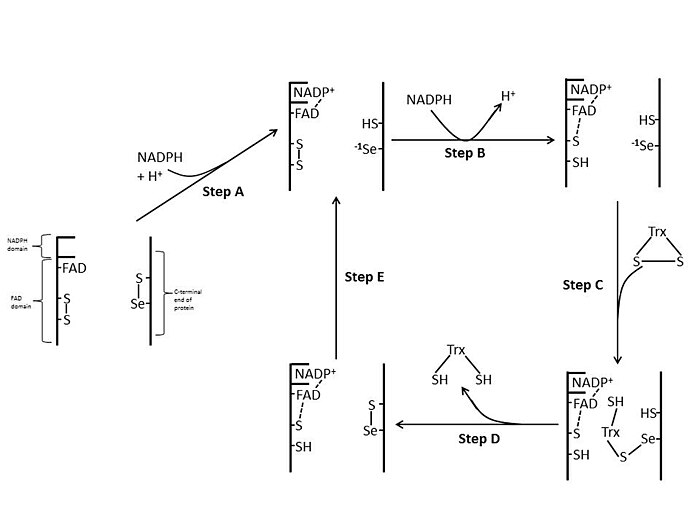
Mamífero
Los TrxR de mamíferos tienen una homología de secuencia mucho mayor con la glutatión reductasa que la E. coli . Los residuos de Cys del sitio activo en el dominio FAD y el dominio NADPH unido están muy próximos eliminando la necesidad de una rotación de 66 grados para la transferencia de electrones que se encuentra en E. coli . Una característica adicional del mecanismo de los mamíferos es la presencia de un residuo de selenocisteína en el extremo C-terminal de la proteína que se requiere para la actividad catalítica. Los residuos conservados en el sitio activo de mamíferos son -Cys-Val-Asn-Val-Gly-Cys-.
Métodos de detección
La tiorredoxina reductasa se puede cuantificar mediante varios métodos, como el ensayo DTNB, utilizando el reactivo de Ellman . La serie TRFS de sondas fluorescentes basadas en disulfuro ha demostrado una detección selectiva de TrxR. Mafireyi sintetizó la primera sonda de diselenida que se aplicó en la detección de TrxR. Otros métodos de detección incluyen técnicas inmunológicas y el ensayo de selenocistina-tiorredoxina reductasa (ensayo SC-TR).
Significación clínica
Tratamiento para el cáncer
Dado que la actividad de esta enzima es esencial para el crecimiento y la supervivencia celular, es un buen objetivo para la terapia antitumoral. Además, la enzima está regulada positivamente en varios tipos de cáncer, incluido el mesotelioma maligno . Por ejemplo, la motexafina gadolinio (MGd) es un nuevo agente quimioterapéutico que se dirige selectivamente a las células tumorales, lo que conduce a la muerte celular y la apoptosis mediante la inhibición de la tiorredoxina reductasa y la ribonucleótido reductasa .
Miocardiopatía
La miocardiopatía dilatada ( MCD ) es un diagnóstico común en casos de insuficiencia cardíaca congestiva . Las tiorredoxina reductasas son proteínas esenciales para regular el equilibrio redox celular y mitigar el daño causado por las especies reactivas de oxígeno generadas a través de la fosforilación oxidativa en las mitocondrias . La inactivación de TrxR2 mitocondrial en ratones da como resultado el adelgazamiento de las paredes del corazón ventricular y la muerte neonatal. Además, se encuentran dos mutaciones en el gen TrxR2 en pacientes diagnosticados con DCM y no en una población de control. Se plantea la hipótesis de que el impacto patológico de estas mutaciones es una capacidad alterada para controlar el daño oxidativo en los miocitos cardíacos .
Antibiótico
Recientemente, se han realizado algunas investigaciones para demostrar que la tiorredoxina reductasa de bajo peso molecular podría ser un objetivo para nuevos antibióticos (como auranofina o Ebselen). Esto es especialmente cierto para Mycobacterium Haemophilum y podría usarse para bacterias resistentes a los antibióticos.
Referencias
enlaces externos
- Thioredoxin + Reductase + (NADPH) en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .