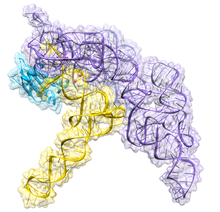
Ribonucleasa P - Ribonuclease P
| RNasa P bacteriana clase A | |
|---|---|
| |
| Identificadores | |
| Símbolo | RNaseP_bact_a |
| Rfam | RF00010 |
| Otros datos | |
| Tipo de ARN | Gene ; ribozima |
| Dominio (s) | Bacterias |
| IR | IR: 0008033 IR: 0004526 IR: 0030680 |
| ASI QUE | Entonces: 0000386 |
| Estructuras PDB | PDBe |
| RNasa bacteriana P clase B | |
|---|---|

| |
| Identificadores | |
| Símbolo | RNaseP_bact_b |
| Rfam | RF00011 |
| Otros datos | |
| Tipo de ARN | Gene ; ribozima |
| Dominio (s) | Bacterias |
| IR | IR: 0008033 IR: 0004526 IR: 0030680 |
| ASI QUE | Entonces: 0000386 |
| Estructuras PDB | PDBe |
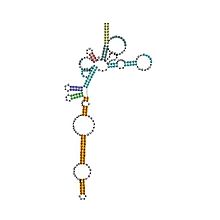
| Archaeal RNase P | |
|---|---|
| |
| Identificadores | |
| Símbolo | RNaseP_arch |
| Rfam | RF00373 |
| Otros datos | |
| Tipo de ARN | Gene ; ribozima |
| Dominio (s) | Arqueas |
| IR | IR: 0008033 IR: 0004526 IR: 0030680 |
| ASI QUE | Entonces: 0000386 |
| Estructuras PDB | PDBe |
| Archaeal RNase P clase T | |
|---|---|
| Identificadores | |
| Símbolo | RNaseP-T |
| Rfam | RF02357 |
| Otros datos | |
| Tipo de ARN | Gene ; ribozima |
| Dominio (s) | Arqueas |
| IR | IR: 0008033 IR: 0004526 IR: 0030680 |
| ASI QUE | Entonces: 0000386 |
| Estructuras PDB | PDBe |
La ribonucleasa P ( EC 3.1.26.5 , RNasa P ) es un tipo de ribonucleasa que escinde el ARN . La RNasa P es única de otras RNasas en que es una ribozima , un ácido ribonucleico que actúa como catalizador de la misma manera que lo haría una enzima basada en proteínas . Su función es escindir una secuencia extra o precursora de ARN en moléculas de ARNt . Además, la RNasa P es una de las dos ribozimas de recambio múltiple conocidas en la naturaleza (la otra es el ribosoma ), cuyo descubrimiento le valió a Sidney Altman y Thomas Cech el Premio Nobel de Química en 1989: en la década de 1970, Altman descubrió la existencia de un precursor ARNt con secuencias flanqueantes y fue el primero en caracterizar la ARNasa P y su actividad en el procesamiento de la secuencia líder 5 ' del ARNt precursor. Los hallazgos recientes también revelan que la RNasa P tiene una nueva función. Se ha demostrado que la ARNasa P nuclear humana es necesaria para la transcripción normal y eficiente de varios ARN pequeños no codificantes , como ARNt, ARNr 5S , ARN SRP y ARNsn U6 , que son transcritos por la ARN polimerasa III , uno de los tres principales genes nucleares. ARN polimerasas en células humanas.
En bacterias
La ARNasa P bacteriana tiene dos componentes: una cadena de ARN, llamada ARN M1, y una cadena polipeptídica, o proteína, llamada proteína C5. In vivo , ambos componentes son necesarios para que la ribozima funcione correctamente, pero in vitro , el ARN M1 puede actuar solo como catalizador. El papel principal de la proteína C5 es mejorar la afinidad de unión al sustrato y la velocidad catalítica de la enzima de ARN M1 probablemente aumentando la afinidad del ión metálico en el sitio activo. La estructura cristalina de una holoenzima de ARNasa P bacteriana con ARNt se ha resuelto recientemente, mostrando cómo los grandes dominios helicoidales apilados coaxialmente del ARN de ARNasa P se involucran en el reconocimiento selectivo de forma de la diana pre-ARNt. Esta estructura cristalina confirma modelos anteriores de reconocimiento y catálisis de sustrato, identifica la ubicación del sitio activo y muestra cómo el componente proteico aumenta la funcionalidad de la RNasa P.
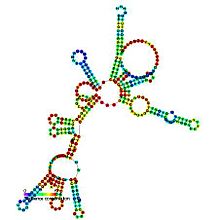
RNasa P bacteriana clase A y B
La ribonucleasa P (RNasa P) es una endoribonucleasa ubicua que se encuentra en arqueas, bacterias y eucarias, así como en cloroplastos y mitocondrias. Su actividad mejor caracterizada es la generación de extremos 5 'maduros de los ARNt mediante la escisión de los elementos líderes en 5' de los ARNt precursores. La RNasa celular Ps son ribonucleoproteínas (RNP). El ARN de la ARNasa Ps bacteriana conserva su actividad catalítica en ausencia de la subunidad proteica, es decir, es una ribozima. No se ha demostrado que el ARN aislado de ARNasa P eucariota y arquea retenga su función catalítica, pero sigue siendo esencial para la actividad catalítica de la holoenzima. Aunque las holoenzimas arqueales y eucariotas tienen un contenido proteico mucho mayor que las eubacterianas, los núcleos de ARN de los tres linajes son homólogos: las hélices correspondientes a P1, P2, P3, P4 y P10 / 11 son comunes a todas las RNasa P celulares. ARN. Sin embargo, existe una considerable variación de secuencia, particularmente entre los ARN eucariotas.
En Archaea
En las arqueas , las ribonucleoproteínas de ARNasa P constan de 4-5 subunidades de proteínas que están asociadas con el ARN. Como revelaron los experimentos de reconstitución in vitro, estas subunidades de proteínas son prescindibles individualmente para el procesamiento del ARNt que está esencialmente mediado por el componente de ARN. Las estructuras de las subunidades de proteínas de la ARNasa P de arqueas se han resuelto mediante cristalografía de rayos X y RMN , revelando así nuevos dominios de proteínas y plegamiento fundamental para la función.
Utilizando genómica comparativa y métodos computacionales mejorados, se ha encontrado una forma radicalmente minimizada del ARN RNasa P, denominada "Tipo T", en todos los genomas completos de la familia filogenética de crenarqueas Thermoproteaceae, incluidas especies de los géneros Pyrobaculum, Caldivirga y Vulcanisaeta. Todos conservan un dominio catalítico convencional, pero carecen de un dominio de especificidad reconocible. Se confirmó experimentalmente la actividad de procesamiento del ARNt 5 'del ARN solo. Los ARN de Pyrobaculum y Caldivirga RNase P son la forma natural más pequeña que se ha descubierto que funciona como ribozimas de acción trans. La pérdida del dominio de especificidad en estos ARN sugiere una posible alteración de la especificidad del sustrato.
Recientemente se ha argumentado que el archaebacteriium Nanoarchaeum equitans no posee RNase P. Los estudios computacionales y experimentales no lograron encontrar evidencia de su existencia. En este organismo, el promotor del ARNt está cerca del gen del ARNt y se cree que la transcripción comienza en la primera base del ARNt, eliminando así el requisito de ARNasa P.
En eucariotas
En eucariotas , como los seres humanos y las levaduras , la mayoría de la ARNasa P consta de una cadena de ARN que es estructuralmente similar a la que se encuentra en las bacterias, así como de nueve a diez proteínas asociadas (a diferencia de la proteína ARNasa P bacteriana única, C5). Cinco de estas subunidades de proteínas exhiben homología con sus contrapartes de arqueas. Estas subunidades proteicas de la ARNasa P se comparten con la ARNasa MRP , una ribonucleoproteína catalítica involucrada en el procesamiento del ARN ribosómico en el nucleolo . Sólo recientemente se demostró que la RNasa P de eucariotas es una ribozima. En consecuencia, las numerosas subunidades proteicas de la RNasa P eucarística tienen una contribución menor al procesamiento del ARNt per se, mientras que parecen ser esenciales para la función de la ARNasa P y la ARNasa MRP en otros entornos biológicos, como la transcripción génica y el ciclo celular . A pesar de los orígenes bacterianos de las mitocondrias y los cloroplastos, los plástidos de animales y plantas superiores no parecen contener una ARNasa P basada en ARN. Se ha demostrado que la ARNasa P mitocondrial humana es una proteína y no contiene ARN . También se ha demostrado que la ARNasa P del cloroplasto de espinaca funciona sin una subunidad de ARN.
| Subunidad | Función / interacción (en el procesamiento de ARNt) |
|---|---|
| RPP14 | Unión de ARN |
| RPP20 | ATPasa, helicasa / Hsp27, SMN, Rpp25 |
| RPP21 | Unión de ARN, actividadg / Rpp29 |
| RPP25 | Unión de ARN / Rpp20 |
| RPP29 | Unión de tRNA, actividad / Rpp21 |
| RPP30 | Unión de ARN, actividad / Pop5 |
| RPP38 | Unión de ARN, actividad |
| RPP40 | |
| hPop1 | |
| hPop5 | Unión de ARN, actividad / Rpp30 |
| ARN H1 | Actividad / Rpp21, Rpp29, Rpp30, Rpp38 |
Terapias que utilizan RNase P
La RNasa P se está estudiando ahora como una terapia potencial para enfermedades como el virus del herpes simple , el citomegalovirus , la influenza y otras infecciones respiratorias, el VIH-1 y el cáncer causado por el gen de fusión BCR-ABL . Las secuencias de guía externas (EGS) se forman con complementariedad con el ARNm viral u oncogénico y las estructuras que imitan el bucle T y el tallo aceptor del ARNt . Estas estructuras permiten que la RNasa P reconozca el EGS y escinda el ARNm diana. Las terapias EGS han demostrado ser efectivas en cultivo y en ratones vivos.
Referencias
Otras lecturas
- Frank DN, Pace NR (1998). "Ribonucleasa P: unidad y diversidad en una ribozima de procesamiento de ARNt" . Revisión anual de bioquímica . 67 : 153–80. doi : 10.1146 / annurev.biochem.67.1.153 . PMID 9759486 .
- Brown JW (enero de 1999). "La base de datos de la ribonucleasa P" . Investigación de ácidos nucleicos . 27 (1): 314. doi : 10.1093 / nar / 27.1.314 . PMC 148169 . PMID 9847214 .
enlaces externos
- Conferencia Nobel de Sidney Altman , premio Nobel de Química 1989
- Base de datos de RNase P en ncsu.edu
- Página de RNasa P nuclear en Rfam
- Página de Archaeal RNase P en Rfam
- Página de RNasa P bacteriana clase A en Rfam
- Página para la clase B de RNasa P bacteriana en Rfam
- RNase + P en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .
- EC 3.1.26.5