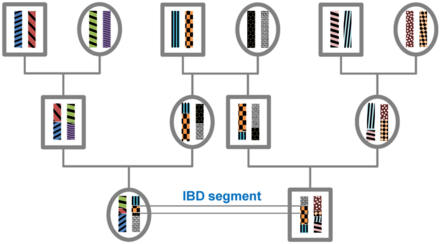
Identidad por descendencia - Identity by descent
Un segmento de ADN es idéntico por estado (IBS) en dos o más individuos si tienen secuencias de nucleótidos idénticas en este segmento. Un segmento de IBS es idéntico por descendencia ( IBD ) en dos o más individuos si lo han heredado de un ancestro común sin recombinación , es decir, el segmento tiene el mismo origen ancestral en estos individuos. Los segmentos de ADN que son IBD son IBS por definición, pero los segmentos que no son IBD aún pueden ser IBS debido a las mismas mutaciones en diferentes individuos o recombinaciones que no alteran el segmento.
Teoría
Todos los individuos de una población finita están relacionados si se remontan lo suficiente y, por lo tanto, compartirán segmentos de sus genomas EII. Durante la meiosis, los segmentos de la EII se rompen por recombinación. Por lo tanto, la longitud esperada de un segmento de IBD depende del número de generaciones desde el ancestro común más reciente en el locus del segmento. La longitud de los segmentos de EII que resultan de un ancestro común n generaciones en el pasado (por lo tanto, involucra 2 n meiosis) se distribuye exponencialmente con una media de 1 / (2 n ) Morgan (M). El número esperado de segmentos de EII disminuye con el número de generaciones desde el ancestro común en este locus. Para un segmento de ADN específico, la probabilidad de ser EII disminuye en 2 -2 n ya que en cada meiosis la probabilidad de transmitir este segmento es 1/2.
Aplicaciones
Los segmentos de IBD identificados se pueden utilizar para una amplia gama de propósitos. Como se señaló anteriormente, la cantidad (duración y número) de la EII que se comparte depende de las relaciones familiares entre los individuos evaluados. Por lo tanto, una aplicación de la detección de segmentos de EII es cuantificar la relación. La medición de parentesco puede usarse en genética forense , pero también puede aumentar la información en el mapeo de ligamiento genético y ayudar a disminuir el sesgo por relaciones indocumentadas en estudios de asociación estándar . Otra aplicación de la EII es la imputación de genotipos y la inferencia de fase de haplotipos . Los segmentos largos compartidos de IBD, que están divididos por regiones cortas, pueden ser indicativos de errores de fase.
Mapeo de EII
El mapeo de EII es similar al análisis de ligamiento, pero se puede realizar sin un pedigrí conocido en una cohorte de individuos no emparentados. El mapeo de la EII puede verse como una nueva forma de análisis de asociación que aumenta el poder de mapear genes o regiones genómicas que contienen múltiples variantes de susceptibilidad a enfermedades raras.
Utilizando datos simulados, Browning y Thompson demostraron que el mapeo de la EII tiene mayor poder que las pruebas de asociación cuando múltiples variantes raras dentro de un gen contribuyen a la susceptibilidad a la enfermedad. A través del mapeo de la EII, se encontraron regiones significativas en todo el genoma en poblaciones aisladas, así como en poblaciones exógamas, mientras que las pruebas de asociación estándar fallaron. Houwen y col. utilizó el intercambio de EII para identificar la ubicación cromosómica de un gen responsable de la colestasis intrahepática recurrente benigna en una población pesquera aislada. Kenny y col. también usó una población aislada para mapear con precisión una señal encontrada por un estudio de asociación de todo el genoma (GWAS) de los niveles de esterol vegetal en plasma (PPS), una medida sustituta de la absorción de colesterol en el intestino. Francks y col. pudo identificar un locus de susceptibilidad potencial para la esquizofrenia y el trastorno bipolar con datos de genotipo de muestras de casos y controles. Lin y col. encontró una señal de vinculación significativa en todo el genoma en un conjunto de datos de pacientes con esclerosis múltiple . Letouzé y col. utilizó el mapeo de la EII para buscar mutaciones fundadoras en muestras de cáncer .
EII en genética de poblaciones
La detección de la selección natural en el genoma humano también es posible mediante segmentos de IBD detectados. Por lo general, la selección tenderá a aumentar el número de segmentos de EII entre los individuos de una población. Mediante la exploración de regiones con un exceso de distribución de EII, se pueden identificar las regiones del genoma humano que han estado sometidas a una selección fuerte y muy reciente.
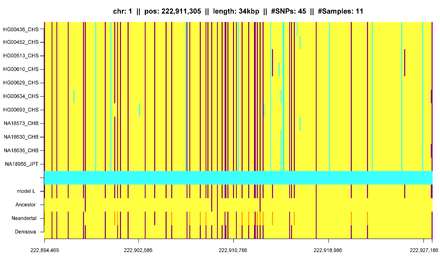
Además de eso, los segmentos de EII pueden ser útiles para medir e identificar otras influencias en la estructura de la población. Gusev y col. mostró que los segmentos de EII se pueden utilizar con modelos adicionales para estimar el historial demográfico, incluidos los cuellos de botella y la mezcla . Utilizando modelos similares Palamara et al. y Carmi et al. reconstruyó la historia demográfica de los judíos asquenazíes y los masai de Kenia . Botigué et al. investigó las diferencias en la ascendencia africana entre las poblaciones europeas. Ralph y Coop utilizaron la detección de EII para cuantificar la ascendencia común de diferentes poblaciones europeas y Gravel et al. De manera similar, trató de sacar conclusiones de la historia genética de las poblaciones de las Américas. Ringbauer y col. utilizó la estructura geográfica de los segmentos de EII para estimar la dispersión dentro de Europa del Este durante los últimos siglos. Utilizando los datos de 1000 genomas , Hochreiter encontró diferencias en el intercambio de EII entre las poblaciones africanas, asiáticas y europeas, así como en los segmentos de EII que se comparten con genomas antiguos como el neandertal o el denisova .
Métodos y software
Programas para la detección de segmentos de EII en personas no relacionadas:
- RAPID : Identidad ultrarrápida por detección de descenso en cohortes a escala de biobanco utilizando la transformada posicional Burrows-Wheeler
- Parente : identifica segmentos de EII entre pares de individuos en datos de genotipo sin fase
- BEAGLE / fastIBD : encuentra segmentos de EII entre pares de individuos en datos de SNP de todo el genoma
- BEAGLE / RefinedIBD : encuentra segmentos de EII en pares de individuos usando un método de hash y evalúa su importancia a través de una razón de verosimilitud
- IBDseq : detecta segmentos de IBD por pares en los datos de secuenciación
- GERMLINE : descubre segmentos de EII en tiempo lineal en parejas de individuos
- DASH : se basa en segmentos de IBD por pares para inferir grupos de individuos que probablemente compartan un solo haplotipo
- PLINK : es un conjunto de herramientas para la asociación del genoma completo y los análisis de vinculación basados en la población que incluye un método para la detección de segmentos de EII por pares.
- Relacionar : estima la probabilidad de EII entre pares de individuos en un locus específico usando SNP
- MCMC_IBDfinder : se basa en la cadena de Markov Monte Carlo (MCMC) para encontrar segmentos de EII en varios individuos
- IBD-Groupon : detecta segmentos de IBD por grupo según las relaciones de IBD por pares
- HapFABIA : identifica segmentos de EII muy cortos caracterizados por variantes raras en grandes datos de secuenciación simultáneamente en varios individuos
Ver también
- Mapeo de asociaciones
- Asociación genética
- Ligamiento genético
- Estudio de asociación de genoma completo
- Identidad por tipo
- Desequilibrio de ligamiento
- Genética de poblaciones