NeuronStudio - NeuronStudio
NeuronStudio fue un programa no comercial creado en Icahn School of Medicine en Mount Sinai por el Computational Neurobiology and Imaging Center. Este programa realizó el rastreo automático y la reconstrucción de estructuras neuronales a partir de pilas de imágenes confocales . Los modelos resultantes se exportaron luego a un archivo utilizando formatos estándar para su posterior procesamiento, modelado o análisis estadísticos. NeuronStudio manejó los detalles morfológicos en escalas que abarcan la geometría de la columna dendrítica local a través de una topología de árbol compleja hasta la disposición espacial general de las redes de múltiples neuronas. Su capacidad de digitalización automatizada evitó los errores subjetivos inherentes al rastreo manual. El programa dejó de recibir apoyo en 2012 y las páginas del proyecto finalmente se eliminaron del sitio web del ISMMS. Sin embargo, su documentación y el código fuente de Windows todavía están disponibles a través de Internet Archive .
Deconvolución
La desconvolución de los datos de las imágenes es esencial para realizar reconstrucciones 3D precisas. La deconvolución es un enfoque de restauración de imágenes en el que se utiliza el conocimiento "a priori" del sistema óptico en forma de función de dispersión de puntos (PSF) para obtener una mejor estimación del objeto. Una función de dispersión puntual puede calcularse a partir de los parámetros reales del microscopio, medirse con perlas o estimarse y refinarse iterativamente ( deconvolución ciega ). Las PSF se pueden ajustar localmente para tener en cuenta las variaciones en las características de refracción del tejido con las características de profundidad y muestra. Para el uso automatizado con grandes bloques de tejido en mosaico, esto es más rápido y más preciso que usar una PSF determinada experimentalmente .
Esqueletización y estimación de diámetro
Los errores de cuantificación surgen en los algoritmos de esqueletización estándar debido a la naturaleza entera de las imágenes digitales. El requisito de una representación precisa de la geometría dendrítica fina ha requerido el desarrollo de nuevas adaptaciones de esqueletización estándar y algoritmos de estimación de diámetro para corregir estos errores de cuantificación. Los métodos iterativos de esqueletización de adelgazamiento pueden proporcionar una distancia en vóxeles desde cada nodo del árbol hasta la superficie del objeto. Esta distancia es la métrica D6, obtenida contando el número de vóxeles a medida que se eliminan en la ruta mínima de 6 conectados desde la superficie hasta el eje medial. En los algoritmos de esqueletización o vectorización existentes para la morfometría dendrítica, la sección transversal de la rama en cualquier nodo se aproxima como circular, con la métrica D6 que proporciona la estimación del diámetro único. La precisión de esta estimación de diámetro se limita al tamaño físico de los vóxeles. Para estructuras pequeñas como dendritas delgadas y espinas, que comprenden solo unos pocos vóxeles incluso con la máxima resolución de imagen, el error puede ser significativo si esta medida se usa directamente (ver figura). Para minimizar el error de cuantificación y evaluar con mayor precisión la geometría de los nodos, existe una nueva técnica de estimación, el Algoritmo de muestreo Rayburst que utiliza los datos originales en escala de grises en lugar de las imágenes segmentadas para una estimación de radio continua y precisa, y muestreo de radio multidireccional para representar con mayor precisión secciones transversales de ramas no circulares y cabezas de espina no esféricas.
Algoritmo de Rayburst
El algoritmo de muestreo de Rayburst utiliza los datos originales en escala de grises en lugar de las imágenes segmentadas para una estimación de radio continua y precisa, y un muestreo de radio multidireccional para representar con mayor precisión secciones transversales de ramas no circulares y cabezas de espinas no esféricas. El algoritmo calcula previamente una matriz de vectores unitarios que muestrean los datos en múltiples direcciones (el núcleo de muestreo) a partir de los cuales se calcula una estimación de la geometría del nodo. La representación precisa de cada dirección por parte del núcleo de muestreo requiere que los N vectores estén espaciados uniformemente sobre la esfera unitaria. El algoritmo utiliza una simulación de física de partículas en la que se genera un conjunto N de vectores unitarios orientados aleatoriamente, lo que da como resultado una distribución aleatoria no uniforme de puntos en la esfera. Luego, cada partícula recibe una fuerza repulsiva de todas las demás partículas, proporcional al cuadrado inverso de la distancia entre ellas. Al desplazar iterativamente la partícula en la dirección de las fuerzas resultantes, las partículas se reorganizan. Este sistema tenderá a una configuración de energía mínima estable dentro de aproximadamente 40 iteraciones, donde cada partícula está separada al máximo de sus vecinas más cercanas. La información se puede utilizar para reconstruir ramas 3D de formas arbitrariamente irregulares. El diámetro de una sección transversal circular equivalente se calcula en el plano normal al eje medial utilizando el diámetro medio de la banda inferior (MLBD). Para calcular el MLBD, tome el conjunto de muestras y agregue los pares correspondientes de vectores opuestos. Ordene los vectores por tamaño, defina la banda inferior como el 50% inferior y utilice la distancia en la posición N / 4, que representa la mediana de la banda inferior, para estimar el diámetro.
Usos de NeuronStudio
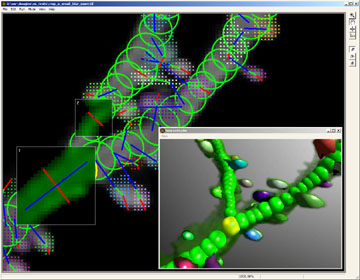
NeuronStudio se utiliza principalmente para el análisis morfológico cuantitativo de las espinas dendríticas . La mayor ventaja de utilizar el rastreo de dendritas y espinas basado en NeuronStudio es que los métodos basados en el rastreo automático no son sensibles a la deriva o el movimiento de las dendritas, porque cada dendrita se puede rastrear por separado en cada pila. Esto permite comparar espinas de grabaciones muy largas o puntos de tiempo más separados. Como el análisis de NeuronStudio da como resultado mediciones 3D precisas del ancho de las cabezas de la columna y la longitud de la columna, este método es útil para detectar cambios en la morfología de la columna. Además, el uso de mediciones por computadora permite el análisis de cientos de espinas en un tiempo relativamente corto.
Ver también
enlaces externos
- Rodríguez, Alfredo (18 de febrero de 2010). "Herramientas: NeuronStudio (Beta) versión 0.9.92, última actualización el 19 de noviembre de 2009" . CNIC, Facultad de Medicina de Mount Sinai. Archivado desde el original el 16 de septiembre de 2018.
-
"Código fuente de NeuronStudio 0.9.92" . CNIC, Facultad de Medicina de Mount Sinai. 2010-09-30. Archivado desde el original el 3 de julio de 2018.
Se permiten trabajos derivados (software basado en partes de este código fuente) siempre que se dé crédito [al autor ] en la publicación y documentación de dichos trabajos.
Referencias
- ^ Rodríguez, A .; Ehlenberger, D .; Kelliher, K .; Einstein, M .; Henderson, SC; Morrison, JH; Hof, PR; Wearne, SL (2003). "Reconstrucción automatizada de la morfología neuronal tridimensional a partir de imágenes de microscopía de barrido láser". Métodos . 30 (1): 94-105. doi : 10.1016 / s1046-2023 (03) 00011-2 . PMID 12695107 .
- ^ Wearne, SL; Rodríguez, A .; Ehlenberger, DB; Rocher, AB; Henderson, SC; Hof, PR (2005). "Nuevas técnicas de imagen, digitalización y análisis de morfología neuronal tridimensional a múltiples escalas" . Neurociencia . 136 (3): 661–680. doi : 10.1016 / j . neurociencia.2005.05.053 . PMID 16344143 .
- ^ Rodríguez, A .; Ehlenberger, D .; Hof, PR; Wearne, SL (2006). "Muestreo Rayburst, un algoritmo para el análisis automatizado de formas tridimensionales a partir de imágenes de microscopía de escaneo láser". Protocolos de la naturaleza . 1 (4): 2156–2161. doi : 10.1038 / nprot.2006.313 . PMID 17487207 .
- ^ Bertling, Enni; Ludwig, Anastasia; Koskinen, Mikko; Hotulainen, Pirta (2012). "Métodos para el análisis tridimensional de la dinámica de la columna dendrítica". En Conn, P. Michael (ed.). Imágenes y análisis espectroscópico de células vivas: imágenes de células vivas en la salud y la enfermedad (PDF) . Métodos en enzimología. 506 . Elsevier. doi : 10.1016 / B978-0-12-391856-7.00043-3 . ISBN 978-0-12-391856-7. ISSN 0076-6879 . Consultado el 14 de julio de 2021 .