HLA-DR - HLA-DR
| MHC clase II , DR | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| (heterodímero) | |||||||||||||||||||
|
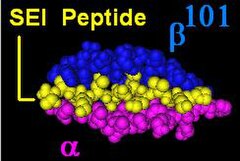
Ilustración de RD con ligando unido (amarillo)
| |||||||||||||||||||
| Tipo de proteina | receptor de superficie celular | ||||||||||||||||||
| Función | Reconocimiento inmunológico y presentación de antígenos |
||||||||||||||||||
| |||||||||||||||||||
HLA-DR es un receptor de superficie celular MHC de clase II codificado por el complejo de antígeno leucocitario humano en la región del cromosoma 6 6p21.31. El complejo de HLA-DR ( H uman L eukocyte A ntigen - DR isotipo) y el péptido, generalmente entre 9 y 30 amino ácidos de longitud, que constituye un ligando para el receptor de células T (TCR). Los HLA ( antígenos leucocitarios humanos ) se definieron originalmente como antígenos de superficie celular que median la enfermedad de injerto contra huésped . La identificación de estos antígenos ha dado lugar a un mayor éxito y longevidad en el trasplante de órganos.
Los antígenos más responsables de la pérdida del injerto son HLA-DR (primeros seis meses), HLA-B (primeros dos años) y HLA-A (supervivencia a largo plazo). Un buen emparejamiento de estos antígenos entre el huésped y el donante es fundamental para lograr la supervivencia del injerto.
HLA-DR también está involucrado en varias condiciones autoinmunes, susceptibilidad a enfermedades y resistencia a enfermedades. También está estrechamente relacionado con HLA-DQ y este vínculo a menudo dificulta la resolución del factor más causante de la enfermedad.
Las moléculas de HLA-DR se regulan positivamente en respuesta a la señalización. En el caso de una infección, el péptido (tal como el péptido de enterotoxina I estafilocócica) se une a una molécula de DR y se presenta a algunos de los muchos receptores de células T que se encuentran en las células T colaboradoras. Estas células luego se unen a antígenos en la superficie de las células B estimulando la proliferación de células B.
Función
La función principal de HLA-DR es presentar antígenos peptídicos, de origen potencialmente extraño, al sistema inmunológico con el fin de provocar o suprimir las respuestas de las células T (auxiliares) que eventualmente conducen a la producción de anticuerpos contra el mismo antígeno peptídico. . Las células presentadoras de antígenos (macrófagos, células B y células dendríticas ) son las células en las que normalmente se encuentra la RD. El aumento de la abundancia de "antígeno" de DR en la superficie celular es a menudo en respuesta a la estimulación y, por lo tanto, la DR también es un marcador de estimulación inmunológica.
Estructura
HLA-DR es un heterodímero αβ , receptor de superficie celular , cada subunidad del cual contiene dos dominios extracelulares, un dominio que atraviesa la membrana y una cola citoplasmática. Tanto las cadenas α como las β están ancladas en la membrana. El dominio N-terminal de la proteína madura forma una hélice alfa que constituye la parte expuesta del surco de unión, la región citoplasmática C-terminal interactúa con la otra cadena formando una hoja beta debajo del surco de unión que se extiende a la membrana celular. La mayoría de las posiciones de contacto del péptido se encuentran en los primeros 80 residuos de cada cadena.
Genética
La genética de HLA-DR es compleja. HLA-DR está codificado por varios loci y varios "genes" de función diferente en cada locus. La cadena α de DR está codificada por el locus HLA-DRA . A diferencia de los otros loci DR, la variación funcional en los productos del gen DRA maduro está ausente. (Nota: consulte la tabla Número de alelos variantes HLA-DR Loci : reduce las posibles combinaciones funcionales de ~ 1400 a ~ 400 ([la tabla no es exacta porque se agregan continuamente nuevos alelos; no todos los alelos nuevos son variantes funcionales de las subunidades maduras ]).
| DR | DR - DQ | DR | DQ | Frec | |||
|---|---|---|---|---|---|---|---|
| Serotipo | haplotipo | B1 | A1 | B1 | % | ||
| DR1 | DR1 - DQ5 | 01:01 | 01:01 | 05:01 | 9. | 1 | |
| 01:02 | 01:01 | 05:01 | 1. | 4 | |||
| 01:03 | 01:01 | 05:01 | 0. | 5 | |||
| DR3 | DR3 - DQ2 | 03:01 | 05:01 | 02:01 | 13. | 1 | |
| DR4 | DR4 - DQ7 | 04:01 | 0300 | 03:01 | 5. | 4 | |
| 04:07 | 0300 | 03:01 | 0. | 9 | |||
| DR4 - DQ8 | 04:01 | 0300 | 03:02 | 5. | 0 | ||
| 04:02 | 0300 | 03:02 | 1. | 0 | |||
| 04:03 | 0300 | 03:02 | 0. | 4 | |||
| 04:04 | 0300 | 03:02 | 3. | 9 | |||
| 04:05 | 0300 | 03:02 | 0. | 3 | |||
| DR7 | DR7 - DQ2 | 07:01 | 02:01 | 02:02 | 11. | 1 | |
| DR7 - DQ9 | 07:01 | 02:01 | 03:03 | 3. | 7 | ||
| DR8 | DR8 - DQ4 | 08:01 | 04:01 | 04:02 | 2. | 2 | |
| DR8 - DQ7 | 08:03 | 06:01 | 03:01 | 0. | 1 | ||
| DR9 | DR9 - DQ9 | 09:01 | 0300 | 03:03 | 0. | 8 | |
| DR10 | DR10 - DQ5 | 10:01 | 01:04 | 05:01 | 0. | 7 | |
| DR11 | DR11 - DQ7 | 11:01 | 05:05 | 03:01 | 5. | 6 | |
| 11:03 | 05:05 | 03:01 | 0. | 3 | |||
| 11:04 | 05:05 | 03:01 | 2. | 7 | |||
| DR12 | DR12 - DQ7 | 12:01 | 05:05 | 03:01 | 1. | 1 | |
| DR13 | DR13 - DQ6 | 13:01 | 01:03 | 06:03 | 5. | 6 | |
| 13:02 | 01:02 | 06:04 | 3. | 4 | |||
| 13:02 | 01:02 | 06:09 | 0. | 7 | |||
| DR13 - DQ7 | 13:03 | 05:05 | 03:01 | 0. | 7 | ||
| DR14 | DR14 - DQ5 | 14:01 | 01:04 | 05:03 | 2. | 0 | |
| DR15 | DR15 - DQ6 | 15:01 | 01:02 | 06:02 | 14. | 2 | |
| 15:02 | 01:03 | 06:01 | 0. | 7 | |||
| DR16 | DR16 - DQ5 | 16:01 | 01:02 | 05:02 | 1. | 0 | |
La cadena β de DR está codificada por 4 loci, sin embargo, no más de 3 loci funcionales están presentes en un solo individuo y no más de dos en un solo cromosoma. A veces, un individuo puede poseer solo 2 copias del mismo locus, DRB1 *. El locus HLA-DRB1 es ubicuo y codifica una gran cantidad de productos génicos funcionalmente variables ( HLA-DR1 a HLA-DR17 ). El locus HLA-DRB3 codifica la especificidad de HLA-DR52 , es moderadamente variable y se asocia de forma variable con ciertos tipos de HLA-DRB1 . El locus HLA-DRB4 codifica la especificidad de HLA-DR53 , tiene alguna variación y está asociado con ciertos tipos de HLA-DRB1 . El locus HLA-DRB5 codifica la especificidad HLA-DR51 , que normalmente es invariable y está vinculada a los tipos HLA-DR2 .
- vinculación (ver tabla)
-
DQA1 y DQB1
- Existe un desequilibrio de ligamiento para muchos tipos de DR-DQ .
- Problemas de nomenclatura. Algunos estudios más antiguos pueden referirse a DR15 o 16 como DR2 y DQ5 y DQ6 como DQ1, por lo tanto, un haplotipo DR2-DQ1 generalmente se refiere a DR15-DQ6 pero podría referirse a DR16-DQ5. DR5 se utiliza para hacer referencia a DR11 y DR12, en cuyo caso se podría utilizar DQ3. En estos casos, DQ3 casi siempre se puede interpretar como DQ7, pero DR5 es más a menudo DR11 y menos frecuentemente DR12. Existen problemas similares para DR6 frente a DR13 y DR14. DR6-DQ1 puede referirse a DR13-DQ6 o con menor frecuencia a DR14-DQ5, pero DR6-DQ3 o DR6-DQ7 generalmente se refiere a DR13-DQ7. Incluso la literatura más antigua tiene designaciones más confusas. Al observar el cambio de asociación de la enfermedad con la mejora de las pruebas, podemos ver cómo ha evolucionado la nomenclatura HLA con el tiempo.
-
DQA1 y DQB1
| HLA-DR | ||||
|---|---|---|---|---|
| HLA | - A1 | - B1 | -B3 a -B5 1 | Potencial |
| Lugar | # | # | # | Combinaciones |
| Alelos | 3 | 463 | 74 | 1635 |
| Polipéptido único | 2 | 394 | 57 | 902 |
| Variante de contacto | 1 | ~ 300 | ~ 30 | ~ 330 |
| 1 DRB3, DRB4, DRB5 tienen presencia variable en humanos | ||||
Evolución y frecuencias alélicas
Hay un alto nivel de diversidad alélica en HLA DRB1, solo es superado por el locus HLA-B en número de variantes alélicas. Estos dos loci son la tasa de variación de secuencia más alta dentro del genoma humano. Esto significa que HLA-DRB1 está evolucionando rápidamente, mucho más rápidamente que casi todos los demás loci que codifican proteínas. Gran parte de la variación en HLA DRB1 ocurre en las posiciones de contacto del péptido en el surco de unión, como resultado, muchos de los alelos alteran la forma en que el DR se une a los ligandos peptídicos y cambia el repertorio que cada receptor puede unirse. Esto significa que la mayoría de los cambios son de naturaleza funcional y, por lo tanto, están bajo selección. En la región HLA, los genes están bajo selección heterocigótica o de equilibrio, aunque ciertos alelos parecen estar bajo selección positiva o negativa, ya sea en el pasado o en el presente.
Los HLA generalmente evolucionan a través de un proceso de conversión de genes , que es una forma de recombinación genética a corta distancia o "abortiva" . Los motivos funcionales en los genes se intercambian para formar nuevos alelos y, con frecuencia, nuevas isoformas de DR funcionalmente diferentes . HLA-DR representa un ejemplo extremo de esto. Un estudio de los loci ligados al cromosoma X revela que la mayoría de los loci humanos han sufrido fijación en los últimos 600.000 años, y los loci diploides han sufrido una proporción significativa de fijación en ese período de tiempo.
El nivel de ramificación profunda en los loci ligados al X indica que los loci estaban cerca de la fijación o se fijaron al final del cuello de botella de la población humana hace 100.000 a 150.000 años. El locus HLA-DR representa una excepción importante a esta observación. Sobre la base de la distribución de los grupos principales en la población humana, es posible afirmar que más de una docena de variantes principales sobrevivieron al cuello de botella de la población. Esta observación está respaldada por el concepto de un coeficiente de selección heterocigoto que opera en el HLA-DR y en el locus HLA-DRB1 en mayor grado en relación con HLA-DQB1 y HLA-DPB1 . La mayoría de los alelos HLA actualmente presentes en la población humana pueden explicarse por la conversión de genes entre estos tipos ancestrales antiguos, algunos que persisten en la población existente.
Serogrupos
| Serotipos de productos del gen HLA-DRB1 | ||
| Antígenos divididos | ||
| HLA-DR1 | ||
| HLA-DR2 | HLA-DR15 | HLA-DR16 |
| HLA-DR3 | HLA-DR17 | HLA-DR18 |
| HLA-DR4 | ||
| HLA-DR5 | HLA-DR11 | HLA-DR12 |
| HLA-DR6 | HLA-DR13 | HLA-DR14 |
| HLA-DR7 | ||
| HLA-DR8 | ||
| HLA-DR9 | ||
| HLA-DR10 | ||
La siguiente tabla proporciona enlaces a subpáginas con información sobre distribución, ligamiento genético y asociación de enfermedades para los serogrupos HLA-DR.
Enlace Interlocus DRB
DRB1 está vinculado con otros loci DRB de cuatro formas.
| no DRB1 | antígenos DRB1 ligados | |||
|---|---|---|---|---|
| antígenos | antígenos | |||
| Ninguno | DR1 | DR8 | DR10 | |
| DR51 | DR2 | DR15 | DR16 | |
| DR52 | DR3 | DR17 | DR18 | |
| DR5 | DR11 | DR12 | ||
| DR6 | DR13 | DR14 | ||
| DR53 | DR4 | DR7 | DR8 | DR9 |
| Clase | Enfermedad | DR asociado | 2 | 3 | 4 |
|---|---|---|---|---|---|
| alopecia areata | DR5 | ||||
| anemia | pernicioso | DR15 | |||
| síndrome antifosfolípido , primario | DR5 | DR12 | |||
| aneurisma | arteria coronaria | DR16 | |||
| arteritis | Takayasu's | DR16 | |||
| artritis , reumatoide | juvenil | DR4 | DR5 | DR14 | DR15 |
| pauciarticular, juv. | DR8 | ||||
| Enfermedad de Still | DR12 | ||||
| iritis con juv. artritis | DR12 | ||||
| seropositivo | DR1 | DR4 | DR10 | ||
| con esclerosis sistémica | DR1 | ||||
| inducida por la enfermedad de Lyme | DR4 | ||||
| intolerancia a la tiopronina | DR5 | DR11 | DR12 | ||
| cardiomiopatía | hipertrófico | DR4 | DR17 | ||
| Inducido por T. cruzi | DR4 | DR7 | DR15 | ||
| colitis | Crohn | DR1 | |||
| ulcerativo | DR1 | ||||
| diabetes | juvenil ( tipo 1 ) | DR3 | DR4 | DR17 | DR18 |
| hígado graso ( tipo 2 ) | DR8 | ||||
| encefalomielitis | inducida por la vacuna contra la rabia | DR17 | |||
| encefalopatía | necrosante aguda | DR52 | |||
| epilepsia | infancia | DR5 | |||
| infantil / espasmo | DR17 | ||||
| enfermedad del corazón | reumático | DR16 | |||
| hepatitis | autoinmune | DR2 | DR4 | DR17 | |
| cirrosis biliar primaria | DR2 | DR8 | |||
| tipo crónico C | DR11 | ||||
| liquen plano | DR1 | DR10 | |||
| lupus , | sistémico | DR3 | DR4 | DR52 | |
| inducido por hidralazina | DR4 | ||||
| con síndrome de Sjögren | DR15 | ||||
| linfadenopatía | generalizado | DR5 | |||
| linfoma , | micosis fungoide | DR5 | |||
| melioidosis | DR16 | ||||
| miastenia | gravis | DR3 | DR6 | DR13 | DR14 |
| inducido por penicilamina | DR1 | ||||
| miositis | cuerpo de inclusión inflamatorio | DR17 | DR18 | DR52 | |
| narcolepsia | DR2 | DR12 | |||
| nefritis , | tubulointersticial | DR1 | |||
| nefropatía | Mediada por IgA | DR4 | |||
| síndrome de deficiencia poliglandular | DR5 | ||||
| pénfigo | foliáceo | DR1 | |||
| vulgaris | DR4 | ||||
| soriasis | vulgaris | DR1 | DR7 | ||
| papilomatosis, | respiratorio | DR1 | |||
| sarcoidosis | no crónico | DR17 | DR52 | ||
| esclerosis , | múltiple | DR2 | DR15 | DR53 | |
| "inicio de la pelea" múltiple | DR3 | ||||
| sistémico | DR4 | DR11 | DR16 | DR52 | |
| liquen vulvar | DR12 | ||||
| esquizofrenia | DR1 | ||||
| susceptibilidad | lepra | DR2 | |||
| tuberculosis | DR2 | ||||
| alergia a la ambrosía Ra6 | DR5 | ||||
| asma, sensible a los ácaros | DR11 | ||||
| 2infección secundaria, SIDA | DR3 | ||||
| aspergilosis | DR15 | ||||
| sarcoma de Kaposi | DR5 | ||||
| carcinomas de tiroides | DR8 | DR11 | |||
| cáncer de ovario / cuello uterino | DR10 | DR11 | DR15 | ||
| anafilaxia inducida por uva | DR11 | ||||
| Chlamydia pneumoniae | DR52 | ||||
| tiroiditis | De Hashimoto | DR3 | DR5 | ||
| Tumbas | DR3 | DR17 | DR52 | ||
| uveítis | tubulointersticial | DR1 | |||
| * las referencias se proporcionan en subpáginas vinculadas | |||||
Referencias
Otras lecturas
- Bénichou S, Benmerah A (2003). "La nef del VIH y las proteínas K3 / K5 del virus asociado al sarcoma de Kaposi:" parásitos "de la vía de la endocitosis" . Med Sci (París) . 19 (1): 100–6. doi : 10.1051 / medsci / 2003191100 . PMID 12836198 .
- Tolstrup M, Ostergaard L, Laursen AL, et al. (2004). "Escape de VIH / VIS de la vigilancia inmunológica: centrarse en Nef". Curr. HIV Res . 2 (2): 141–51. doi : 10.2174 / 1570162043484924 . PMID 15078178 .
- Anderson JL, Hope TJ (2005). "Proteínas accesorias del VIH y supervivientes de la célula huésped". Informes actuales sobre el VIH / SIDA . 1 (1): 47–53. doi : 10.1007 / s11904-004-0007-x . PMID 16091223 . S2CID 34731265 .
- Li L, Li HS, Pauza CD y col. (2006). "Funciones de las proteínas auxiliares del VIH-1 en la patogénesis viral y las interacciones huésped-patógeno" . Cell Res . 15 (11-12): 923-34. doi : 10.1038 / sj.cr.7290370 . PMID 16354571 .
- Estufa V, Verhasselt B (2006). "Modelado de efectos de Nef del VIH-1 tímico". Curr. HIV Res . 4 (1): 57–64. doi : 10.2174 / 157016206775197583 . PMID 16454711 .
- Matsushima GK, Itoh-Lindstrom Y, Ting JP (1992). "Activación del gen HLA-DRA en linfocitos T humanos primarios: uso novedoso de TATA y los elementos promotores X e Y" . Mol. Celda. Biol . 12 (12): 5610–9. doi : 10.1128 / MCB.12.12.5610 . PMC 360500 . PMID 1448091 .
- Schaiff WT, Hruska KA, McCourt DW y col. (1992). "HLA-DR se asocia con proteínas de estrés específicas y se retiene en el retículo endoplásmico en células negativas de cadena invariante" . J. Exp. Med . 176 (3): 657–66. doi : 10.1084 / jem.176.3.657 . PMC 2119345 . PMID 1512535 .
- Piatier-Tonneau D, Gastinel LN, Amblard F, et al. (1991). "Interacción de CD4 con antígenos HLA clase II y VIH gp120". Inmunogenética . 34 (2): 121–8. doi : 10.1007 / BF00211424 . PMID 1869305 . S2CID 10116507 .
- Nong Y, Kandil O, Tobin EH y col. (1991). "La proteína del núcleo del VIH p24 inhibe el aumento inducido por interferón-gamma de los niveles de ARNm de cadena pesada de HLA-DR y del citocromo b en la línea celular THP1 de tipo monocito humano". Celda. Immunol . 132 (1): 10–6. doi : 10.1016 / 0008-8749 (91) 90002-S . PMID 1905983 .
- Rosenstein Y, Burakoff SJ, Herrmann SH (1990). "HIV-gp120 puede bloquear la adhesión mediada por MHC de clase II de CD4". J. Immunol . 144 (2): 526–31. PMID 1967269 .
- Callahan KM, Fort MM, Obah EA y col. (1990). "La variabilidad genética en VIH-1 gp120 afecta las interacciones con moléculas HLA y receptor de células T". J. Immunol . 144 (9): 3341–6. PMID 1970352 .
- Bowman MR, MacFerrin KD, Schreiber SL, Burakoff SJ (1991). "Identificación y análisis estructural de residuos en la región V1 de CD4 implicados en la interacción con la glicoproteína gp120 de la envoltura del virus de inmunodeficiencia humana y moléculas del complejo de histocompatibilidad principal de clase II" . Proc. Natl. Acad. Sci. USA . 87 (22): 9052–6. doi : 10.1073 / pnas.87.22.9052 . PMC 55099 . PMID 1978941 .
- Koppelman B, Cresswell P (1990). "Rápida degradación no lisosomal de glucoproteínas HLA clase II ensambladas que incorporan una cadena alfa de DR mutante". J. Immunol . 145 (8): 2730–6. PMID 2212658 .
- Clayton LK, Sieh M, Pious DA, Reinherz EL (1989). "Identificación de residuos de CD4 humanos que afectan la unión de MHC de clase II frente a la unión de gp120 de VIH-1" . Naturaleza . 339 (6225): 548–51. Código Bibliográfico : 1989Natur.339..548C . doi : 10.1038 / 339548a0 . PMID 2543930 . S2CID 4246781 .
- Diamond DC, Sleckman BP, Gregory T, et al. (1988). "Inhibición de la función de las células T CD4 + por la proteína de la envoltura del VIH, gp120". J. Immunol . 141 (11): 3715–7. PMID 2846691 .
- Tjernlund U, Scheynius A, Johansson C y col. (1989). "Respuesta de células T al derivado de proteína purificada después de la eliminación de células de Langerhans de suspensiones de células epidérmicas que contienen queratinocitos que expresan antígenos de trasplante de clase II". Scand. J. Immunol . 28 (6): 667–73. doi : 10.1111 / j.1365-3083.1988.tb01500.x . PMID 3266023 . S2CID 25824282 .
- Andrieu JM, Even P, Venet A (1986). "SIDA y síndromes relacionados como una enfermedad autoinmune del sistema inmunológico inducida por virus: un trastorno anti-MHC II. Implicaciones terapéuticas". Investigación sobre el SIDA . 2 (3): 163–74. doi : 10.1089 / aid.1.1986.2.163 . PMID 3489470 .
- Das HK, Lawrance SK, Weissman SM (1983). "Estructura y secuencia de nucleótidos del gen de la cadena pesada de HLA-DR" . Proc. Natl. Acad. Sci. USA . 80 (12): 3543–7. Código Bibliográfico : 1983PNAS ... 80.3543D . doi : 10.1073 / pnas.80.12.3543 . PMC 394085 . PMID 6304715 .
- Schamboeck A, Korman AJ, Kamb A, Strominger JL (1984). "Organización de la unidad transcripcional de un antígeno de histocompatibilidad de clase II humana: cadena pesada HLA-DR" . Ácidos nucleicos Res . 11 (24): 8663–75. doi : 10.1093 / nar / 11.24.8663 . PMC 326615 . PMID 6324094 .
- Das HK, Biro PA, Cohen SN y col. (1983). "Uso de sondas de oligonucleótidos sintéticos complementarias a genes para HLA-DR alfa y beta humanos como cebadores de extensión para el aislamiento de clones genómicos específicos de 5 '" . Proc. Natl. Acad. Sci. USA . 80 (6): 1531–5. Código Bibliográfico : 1983PNAS ... 80.1531D . doi : 10.1073 / pnas.80.6.1531 . PMC 393635 . PMID 6403940 .
enlaces externos
- Antígenos HLA-DR + en los títulos de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU .