Biblioteca química codificada por ADN - DNA-encoded chemical library
Las bibliotecas químicas codificadas por ADN ( DEL ) es una tecnología para la síntesis y el cribado a una escala sin precedentes de colecciones de compuestos de moléculas pequeñas . DEL se utiliza en química médica para unir los campos de la química combinatoria y la biología molecular . El objetivo de la tecnología DEL es acelerar el proceso de descubrimiento de fármacos y, en particular, las actividades de descubrimiento de fase temprana como la validación de objetivos y la identificación de aciertos.
La tecnología DEL implica la conjugación de compuestos químicos o bloques de construcción para fragmentos cortos de ADN que sirven como códigos de barras de identificación y, en algunos casos, también dirigen y controlan la síntesis química. La técnica permite la creación masiva y el interrogatorio de bibliotecas mediante selección por afinidad, típicamente en una proteína diana inmovilizada. Recientemente se ha desarrollado un método homogéneo para cribar bibliotecas codificadas por ADN que utiliza tecnología de emulsión de agua en aceite para aislar, contar e identificar complejos ligando-objetivo individuales en un enfoque de un solo tubo. A diferencia de los procedimientos de cribado convencionales, como el cribado de alto rendimiento , no se requieren ensayos bioquímicos para la identificación de aglutinantes, lo que en principio permite el aislamiento de aglutinantes en una amplia gama de proteínas históricamente difíciles de abordar con tecnologías de cribado convencionales. Por lo tanto, además del descubrimiento general de compuestos moleculares específicos diana, la disponibilidad de aglutinantes para proteínas diana farmacológicamente importantes, pero hasta ahora "no farmacológicas", abre nuevas posibilidades para desarrollar fármacos novedosos para enfermedades que hasta ahora no podían tratarse. Al eliminar el requisito de evaluar inicialmente la actividad de los hits, se espera y se espera que muchos de los aglutinantes de alta afinidad identificados se muestren activos en el análisis independiente de los hits seleccionados, ofreciendo así un método eficiente para identificar hits de alta calidad y clientes potenciales farmacéuticos. .
Bibliotecas químicas codificadas por ADN y tecnologías de visualización
Hasta hace poco, la aplicación de la evolución molecular en el laboratorio se había limitado a tecnologías de exhibición que involucraban moléculas biológicas, donde las moléculas pequeñas lideran el descubrimiento se consideraba más allá de este enfoque biológico. DEL ha abierto el campo de la tecnología de visualización para incluir compuestos no naturales como moléculas pequeñas, extendiendo la aplicación de la evolución molecular y la selección natural a la identificación de compuestos de moléculas pequeñas con la actividad y función deseadas. Las bibliotecas químicas codificadas por ADN se parecen a las tecnologías de presentación biológica, como la tecnología de presentación de fagos de anticuerpos , presentación de levadura , presentación de ARNm y aptámero SELEX . En la presentación de anticuerpos en fagos, los anticuerpos se unen físicamente a partículas de fagos que portan el gen que codifica el anticuerpo unido, lo que equivale a un enlace físico de un " fenotipo " (la proteína) y un " genotipo " (el gen que codifica la proteína). ). Los anticuerpos mostrados en fagos pueden aislarse de grandes bibliotecas de anticuerpos imitando la evolución molecular: a través de rondas de selección (en una proteína diana inmovilizada), amplificación y traducción. En DEL, el enlace de una molécula pequeña a un código identificador de ADN permite la fácil identificación de moléculas de unión. Las bibliotecas DEL se someten a procedimientos de selección por afinidad en una proteína diana inmovilizada de elección, después de lo cual los no aglutinantes se eliminan mediante pasos de lavado, y los aglutinantes pueden posteriormente amplificarse mediante la reacción en cadena de la polimerasa (PCR) e identificarse en virtud de su código de ADN (p. Ej. Secuencia ADN). En las tecnologías DEL basadas en la evolución (ver más abajo) los aciertos pueden enriquecerse aún más realizando rondas de selección, amplificación por PCR y traducción en analogía con los sistemas de presentación biológica, como la presentación de fagos de anticuerpos. Esto hace posible trabajar con bibliotecas mucho más grandes.
Historia
“Sintetizar una mezcla de compuestos de varios componentes en un solo proceso y cribarla también en un solo proceso”. Este es el principio de química combinatoria inventado por el Prof. Furka Á. (Universidad Eötvös Loránd Budapest Hungría) en 1982, y lo describió incluyendo el método de síntesis de bibliotecas combinatorias y el de una estrategia de deconvolución en un documento notariado en el mismo año. Las motivaciones que llevaron a la invención se publicaron en 2002. Las DEL son bibliotecas combinatorias codificadas por ADN (DECL) y el principio combinatorio prevalece claramente en su aplicación.

El concepto de codificación del ADN se describió por primera vez en un artículo teórico de Brenner y Lerner en 1992 en el que se propuso vincular cada molécula de una entidad sintetizada químicamente a una secuencia de oligonucleótidos particular construida en paralelo y utilizar esta etiqueta genética codificante para identificar y enriquecer compuestos activos. En 1993, la primera implementación práctica de este enfoque fue presentada por S. Brenner y K. Janda y de manera similar por el grupo de MA Gallop. Brenner y Janda sugirieron generar miembros de la biblioteca codificados individuales mediante una síntesis combinatoria paralela alterna del compuesto químico heteropolimérico y la secuencia de oligonucleótidos apropiada en la misma perla en una forma basada en "split-&-pool" (ver más abajo).
Dado que el ADN sin protección está restringido a una ventana estrecha de las condiciones de reacción convencionales, hasta finales de la década de 1990 se previeron varias estrategias de codificación alternativas (es decir , marcado de compuestos basados en MS , codificación de péptidos , marcado haloaromático , codificación por aminas secundarias , dispositivos semiconductores . ), principalmente para evitar la incómoda síntesis de ADN en fase sólida y para crear bibliotecas combinatorias fácilmente rastreables con un alto rendimiento. Sin embargo, la amplificabilidad selectiva del ADN facilita enormemente el cribado de bibliotecas y se vuelve indispensable para la codificación de bibliotecas de compuestos orgánicos de este tamaño sin precedentes. En consecuencia, a principios de la década de 2000, la química combinatoria del ADN experimentó un renacimiento.
El comienzo del milenio vio la introducción de varios desarrollos independientes en la tecnología DEL. Estas tecnologías se pueden clasificar en dos categorías generales: tecnologías DEL no basadas en la evolución y tecnologías DEL basadas en la evolución capaces de evolución molecular. La primera categoría se beneficia de la capacidad de utilizar reactivos disponibles en el mercado y, por lo tanto, permite una generación de bibliotecas bastante sencilla. Los impactos se pueden identificar mediante secuenciación de ADN, sin embargo, la traducción del ADN y, por lo tanto, la evolución molecular no es factible con estos métodos. Los enfoques de división y agrupación desarrollados por investigadores de Praecis Pharmaceuticals (ahora propiedad de GlaxoSmithKline), Nuevolution (Copenhague, Dinamarca) y la tecnología ESAC desarrollada en el laboratorio del profesor D. Neri (Instituto de Ciencias Farmacéuticas, Zúrich, Suiza) se incluyen en esta categoría. . La tecnología ESAC se distingue por ser un enfoque combinatorio de autoensamblaje que se asemeja al descubrimiento de hits basado en fragmentos (Fig. 1b). Aquí, el recocido de ADN permite muestrear combinaciones de bloques de construcción discretos, pero no se produce ninguna reacción química entre ellos. Ejemplos de tecnologías DEL basadas en la evolución son el enrutamiento de ADN desarrollado por el profesor DR Halpin y el profesor PB Harbury (Universidad de Stanford, Stanford, CA), síntesis de plantilla de ADN desarrollada por el profesor D. Liu (Universidad de Harvard, Cambridge, MA) y comercializado por Ensemble Therapeutics (Cambridge, MA) y la tecnología YoctoReactor. desarrollado y comercializado por Vipergen (Copenhague, Dinamarca). Estas tecnologías se describen con más detalle a continuación. La síntesis de plantilla de ADN y la tecnología YoctoReactor requieren la conjugación previa de bloques de construcción químicos (BB) a una etiqueta de oligonucleótido de ADN antes del ensamblaje de la biblioteca, por lo que se requiere más trabajo previo antes del ensamblaje de la biblioteca. Además, los BB etiquetados con ADN permiten la generación de un código genético para compuestos sintetizados y es posible la traducción artificial del código genético: es decir, los BB pueden recuperarse mediante el código genético amplificado por PCR y los compuestos de la biblioteca pueden regenerarse. Esto, a su vez, permite aplicar el principio de selección natural y evolución darwiniana a la selección de moléculas pequeñas en analogía directa con los sistemas de presentación biológica; a través de rondas de selección, amplificación y traducción.
Tecnologías no basadas en la evolución
Bibliotecas combinatorias
Las bibliotecas combinatorias son mezclas especiales de compuestos multicomponente que se sintetizan en un solo proceso paso a paso. Se diferencian de la colección de compuestos individuales así como de una serie de compuestos preparados por síntesis paralela. Las bibliotecas combinatorias tienen características importantes.
″ Las mezclas se utilizan en su síntesis. El uso de mezclas asegura la altísima eficiencia del proceso. Ambos reactivos podrían ser mezclas, pero por razones prácticas se utiliza el procedimiento de mezcla dividida: una mezcla se divide en porciones que se acoplan con los BB. Las mezclas son tan importantes que no existe una biblioteca combinatoria sin usar una mezcla en la síntesis, y si se usa una mezcla en un proceso, inevitablemente se forma una biblioteca combinatoria.
″ Los componentes de las bibliotecas deben estar presentes en cantidades molares casi iguales. Para lograr esto lo más fielmente posible, las mezclas se dividen en porciones iguales y, después de la agrupación, se necesita una mezcla completa.
″ Dado que se desconoce la estructura de los componentes, es necesario utilizar métodos de deconvolución en el cribado. Por esta razón, se han desarrollado métodos de codificación. Las moléculas codificantes están unidas a las perlas del soporte sólido que registran los BB acoplados y su secuencia. Uno de estos métodos es la codificación de oligómeros de ADN.
″ Es una característica notable de las bibliotecas combinatorias que toda la mezcla de compuestos se puede cribar en un solo proceso.
Dado que tanto la síntesis como el cribado son procedimientos muy eficientes, el uso de bibliotecas combinatorias en la investigación farmacéutica genera enormes ahorros.
En la síntesis combinatoria en fase sólida, solo se forma un único compuesto en cada perla. Por este motivo, el número de componentes de la biblioteca no puede superar el número de perlas del soporte sólido. Esto significa que el número de componentes de dichas bibliotecas es limitado. Esta restricción fue completamente eliminada por Harbury y Halpin. En su síntesis de DEL, se omite el soporte sólido y los BB se unen directamente a los oligómeros de ADN codificantes. Este nuevo enfoque ayuda a aumentar prácticamente de forma ilimitada el número de componentes de las bibliotecas combinatorias codificadas por ADN (DECL).
Codificación de ADN dividida y agrupada
Con el fin de aplicar la química combinatoria para la síntesis de bibliotecas químicas codificadas por ADN, se siguió un enfoque de Split - & - Pool. Inicialmente, un conjunto de oligonucleótidos ( n ) de ADN únicos, cada uno de los cuales contiene una secuencia codificante específica, se conjuga químicamente con un conjunto correspondiente de pequeñas moléculas orgánicas. En consecuencia, los compuestos conjugados de oligonucleótidos se mezclan ("Pool") y se dividen ("Split") en varios grupos ( m ). En condiciones apropiadas, se acopla un segundo conjunto de bloques de construcción (m) al primero y se introduce enzimáticamente un oligonucleótido adicional que codifica la segunda modificación antes de volver a mezclar. Estos pasos de "dividir y agrupar" se pueden iterar varias veces ( r ) aumentando en cada ronda el tamaño de la biblioteca de una manera combinatoria (es decir, ( n x m ) r ). Alternativamente, se han utilizado ácidos nucleicos peptídicos para codificar bibliotecas preparadas mediante el método de "división y agrupación". Una ventaja de la codificación de PNA es que la química se puede realizar mediante SPPS estándar.
Acoplamiento gradual de fragmentos de ADN codificantes a moléculas orgánicas nacientes

Una estrategia prometedora para la construcción de bibliotecas codificadas por ADN está representada por el uso de bloques de construcción multifuncionales conjugados covalentemente a un oligonucleótido que sirve como una "estructura central" para la síntesis de bibliotecas. En forma de agrupación y división, un conjunto de andamios multifuncionales experimenta reacciones ortogonales con una serie de socios reactivos adecuados. Después de cada paso de reacción, la identidad de la modificación se codifica mediante una adición enzimática de un segmento de ADN a la "estructura central" del ADN original. El uso de aminoácidos protegidos con N unidos covalentemente a un fragmento de ADN permite, después de una etapa de desprotección adecuada, una nueva formación de enlace amida con una serie de ácidos carboxílicos o una aminación reductora con aldehídos . De manera similar, los ácidos dieno carboxílicos usados como armazones para la construcción de bibliotecas en el extremo 5 'del oligonucleótido modificado con amino , podrían someterse a una reacción de Diels-Alder con una variedad de derivados de maleimida . Una vez completada la etapa de reacción deseada, la identidad del resto químico añadido al oligonucleótido se establece mediante el apareamiento de un oligonucleótido parcialmente complementario y mediante una polimerización de ADN de relleno Klenow posterior , produciendo un fragmento de ADN de doble hebra. Las estrategias de síntesis y de codificación descrito anteriormente permiten la construcción fácil de bibliotecas codificadas en el ADN de un tamaño de hasta 10 4 compuestos miembro portador dos conjuntos de “bloques de construcción”. Sin embargo, también se puede considerar la adición escalonada de al menos tres conjuntos independientes de restos químicos a un bloque de construcción del núcleo trifuncional para la construcción y codificación de una biblioteca codificada por ADN muy grande (que comprende hasta 10 6 compuestos) ( Fig. 2 )
Autoensamblaje combinatorio
Bibliotecas químicas de autoensamblaje codificadas

E ncoded S elfo A ssembling C hemical ( ESAC ) las bibliotecas se basan en el principio de que dos sub-bibliotecas de un tamaño de x miembros (por ejemplo, 10 3 ) que contiene un dominio de hibridación complementario constante puede producir una biblioteca de ADN combinatoria-duplex después de la hibridación con una complejidad de x 2 miembros de la biblioteca representados uniformemente (por ejemplo, 10 6 ). Cada miembro de la subbiblioteca consistiría en un oligonucleótido que contiene una región codificante variable flanqueada por una secuencia de ADN constante, que lleva una modificación química adecuada en el extremo del oligonucleótido. Las subbibliotecas de ESAC se pueden usar en al menos cuatro realizaciones diferentes.
- Una subbiblioteca se puede emparejar con un oligonucleótido complementario y usarse como una biblioteca codificada por ADN que muestra un único compuesto unido covalentemente para experimentos de selección basados en afinidad.
- Una subbiblioteca se puede emparejar con un oligonucleótido que muestra un aglutinante conocido al objetivo, lo que permite estrategias de maduración por afinidad.
- Se pueden ensamblar dos subbibliotecas individuales de forma combinatoria y usar para la identificación de novo de moléculas de unión bindentadas.
- Se pueden ensamblar tres sub-bibliotecas diferentes para formar una biblioteca tríplex combinatoria.
Los aglutinantes preferenciales aislados de una selección basada en afinidad pueden amplificarse por PCR y decodificarse en micromatrices de oligonucleótidos complementarios o mediante concatenación de los códigos, subclonación y secuenciación . Los bloques de construcción individuales se pueden conjugar eventualmente usando enlazadores adecuados para producir un compuesto de alta afinidad similar a un fármaco. Las características del enlazador (por ejemplo, longitud, flexibilidad, geometría, naturaleza química y solubilidad) influyen en la afinidad de unión y las propiedades químicas del ligante resultante ( figura 3 ).
Los experimentos de biopanning en HSA de una biblioteca ESAC de 600 miembros permitieron el aislamiento del resto 4- ( p- yodofenil) butanoico. El compuesto representa la estructura central de una serie de moléculas de unión a albúmina portátiles y de Albufluor, un agente de contraste angiográfico con fluoresceína desarrollado recientemente , actualmente en evaluación clínica.
La tecnología ESAC se ha utilizado para el aislamiento de potentes inhibidores de la tripsina bovina y para la identificación de nuevos inhibidores de la estromelisina-1 ( MMP-3 ), una metaloproteinasa de matriz implicada en procesos de remodelación tisular tanto fisiológicos como patológicos, así como en procesos patológicos. , como artritis y metástasis .
Tecnologías basadas en la evolución
Enrutamiento de ADN
En 2004, DR Halpin y PB Harbury presentaron un método novedoso e intrigante para la construcción de bibliotecas codificadas por ADN. Por primera vez, las plantillas conjugadas con ADN sirvieron tanto para codificar como para programar la infraestructura de la síntesis de "división y agrupación" de los componentes de la biblioteca. El diseño de Halpin y Harbury permitió rondas alternas de selección, amplificación por PCR y diversificación con pequeñas moléculas orgánicas, en completa analogía con la tecnología de presentación de fagos . La maquinaria de enrutamiento del ADN consta de una serie de columnas conectadas que llevan anticodones unidos a resina, que podrían secuenciar, específicamente, separar una población de plantillas de ADN en ubicaciones espacialmente distintas mediante hibridación . De acuerdo con este protocolo de división y piscina un péptido biblioteca combinatoria de ADN codificada de 10 6 fue generado miembros.
Síntesis de plantilla de ADN

En 2001, David Liu y sus colaboradores demostraron que se pueden usar oligonucleótidos de ADN complementarios para ayudar a ciertas reacciones sintéticas , que no tienen lugar de manera eficiente en solución a baja concentración . Se utilizó un heterodúplex de ADN para acelerar la reacción entre los restos químicos que se muestran en los extremos de las dos cadenas de ADN. Además, se demostró que el "efecto de proximidad", que acelera la reacción bimolecular, es independiente de la distancia (al menos dentro de una distancia de 30 nucleótidos ). De una manera secuencial programada, los oligonucleótidos que llevaban un grupo reactivo químico se hibridaron con derivados oligonucleotídicos complementarios que llevaban un grupo químico reactivo diferente. La proximidad conferida por la hibridación del ADN aumenta drásticamente la molaridad efectiva de los reactivos de reacción unidos a los oligonucleótidos, permitiendo que la reacción deseada ocurra incluso en un ambiente acuoso a concentraciones que son varios órdenes de magnitud inferiores a las necesarias para la correspondiente reacción orgánica convencional. no con plantilla de ADN. Usando una configuración de plantilla de ADN y una síntesis programada por secuencia, Liu y sus colaboradores generaron una biblioteca de macrociclos codificada por ADN compuesto de 64 miembros .
Tecnología basada en proximidad tridimensional (tecnología YoctoReactor)
El YoctoReactor (yR) es un enfoque impulsado por la proximidad 3D que explota la naturaleza de autoensamblaje de los oligonucleótidos de ADN en uniones de 3, 4 o 5 vías para dirigir la síntesis de moléculas pequeñas en el centro de la unión. La Figura 5 ilustra el concepto básico con una unión de ADN de 4 vías.
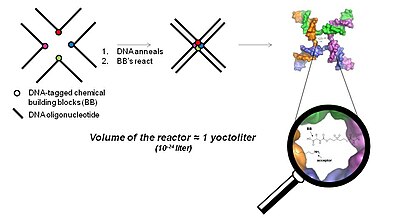
El centro de la unión del ADN constituye un volumen del orden de un yoctolitro , de ahí el nombre YoctoReactor. Este volumen contiene una reacción de una sola molécula que produce concentraciones de reacción en el rango alto de mM. La concentración efectiva facilitada por el ADN acelera enormemente las reacciones químicas que de otro modo no tendrían lugar a la concentración real varios órdenes de magnitud más baja.
Construyendo una biblioteca de año
La Figura 6 ilustra la generación de una biblioteca yR usando una unión de ADN de 3 vías.
En resumen, los bloques de construcción químicos (BB) se unen mediante enlazadores escindibles o no escindibles a tres tipos de oligonucleótidos de ADN biespecíficos (oligo-BB) que representan cada brazo del yR. Para facilitar la síntesis de manera combinatoria, los oligo-BB están diseñados de manera que el ADN contenga (a) el código de un BB adherido en el extremo distal del oligo (líneas de color) y (b) áreas de secuencia constante de ADN (negro líneas) para provocar el autoensamblaje del ADN en una unión de 3 vías (independientemente del BB) y la posterior reacción química. Las reacciones químicas se llevan a cabo mediante un procedimiento paso a paso y después de cada paso, el ADN se liga y el producto se purifica mediante electroforesis en gel de poliacriamida. Los enlazadores escindibles (BB-DNA) se utilizan para todas las posiciones menos una, lo que produce una biblioteca de moléculas pequeñas con un solo enlace covalente al código de ADN. La Tabla 1 describe cómo se pueden generar bibliotecas de diferentes tamaños utilizando la tecnología yR.
El enfoque de diseño yR proporciona un sitio de reacción invariable con respecto a (a) la distancia entre los reactivos y (b) el entorno de secuencia que rodea el sitio de reacción. Además, la conexión íntima entre el código y el BB en los restos oligo-BB que se mezclan combinatoriamente en un solo recipiente confiere una alta fidelidad a la codificación de la biblioteca. El código de los productos sintetizados, además, no está preestablecido, sino que se ensambla combinatoriamente y se sintetiza en sincronía con el producto innato.
Cribado homogéneo de bibliotecas de yoctoreactores
Recientemente se ha desarrollado un método homogéneo para cribar bibliotecas de yoctoreactores (yR) que utiliza tecnología de emulsión de agua en aceite para aislar complejos ligando-objetivo individuales. Denominado Binder Trap Enrichment (BTE), los ligandos de una proteína diana se identifican atrapando pares de unión (proteína diana marcada con ADN y ligando yR) en gotitas de emulsión durante la cinética dominada por la disociación. Una vez atrapados, el ADN del ligando y el objetivo se unen mediante ligación, preservando así la información de unión.
De ahora en adelante, la identificación de aciertos es esencialmente un ejercicio de conteo: la información sobre los eventos de unión se descifra secuenciando y contando el ADN unido; los ligantes selectivos se cuentan con una frecuencia mucho más alta que los ligantes aleatorios. Esto es posible porque la captura aleatoria de la diana y el ligando se "diluye" por el alto número de gotas de agua en la emulsión. La característica de bajo ruido y señal de fondo de BTE se atribuye a la "dilución" de la señal aleatoria, la falta de artefactos superficiales y la alta fidelidad de la biblioteca yR y el método de cribado. El cribado se realiza en un método de un solo tubo. Los aciertos biológicamente activos se identifican en una única ronda de BTE caracterizada por una baja tasa de falsos positivos.
BTE imita la naturaleza de no equilibrio de las interacciones ligando-objetivo in vivo y ofrece la posibilidad única de cribar ligandos específicos objetivo en función del tiempo de residencia ligando-objetivo porque la emulsión, que atrapa el complejo de unión, se forma durante una fase de disociación dinámica.
Decodificación de bibliotecas químicas codificadas por ADN
Tras la selección de bibliotecas químicas codificadas por ADN, la estrategia de decodificación para la identificación rápida y eficiente de los compuestos de unión específicos es crucial para el desarrollo posterior de la tecnología DEL . Hasta ahora, la decodificación basada en secuenciación de Sanger , la metodología basada en microarrays y las técnicas de secuenciación de alto rendimiento representaron las principales metodologías para la decodificación de selecciones de bibliotecas codificadas por ADN.
Decodificación basada en secuenciación de Sanger
Aunque muchos autores previeron implícitamente una decodificación tradicional basada en secuenciación de Sanger , el número de códigos a secuenciar simplemente de acuerdo con la complejidad de la biblioteca es definitivamente una tarea poco realista para un enfoque de secuenciación tradicional de Sanger . Sin embargo, la implementación de la secuenciación de Sanger para decodificar bibliotecas químicas codificadas por ADN en una forma de alto rendimiento fue la primera en ser descrita. Después de la selección y amplificación por PCR de las etiquetas de ADN de los compuestos de la biblioteca, se generaron concatámeros que contenían múltiples secuencias codificantes y se ligaron en un vector . Después de la secuenciación de Sanger de un número representativo de las colonias resultantes , se revelaron las frecuencias de los códigos presentes en la muestra de la biblioteca codificada por ADN antes y después de la selección.
Decodificación basada en microarrays
Una micromatriz de ADN es un dispositivo para investigaciones de alto rendimiento ampliamente utilizado en biología molecular y en medicina . Consiste en una serie ordenada de manchas microscópicas ("características" o "ubicaciones") que contienen pocos picomoles de oligonucleótidos que llevan una secuencia de ADN específica. Puede ser una sección corta de un gen u otro elemento de ADN que se utilizan como sondas para hibridar una muestra de ADN o ARN en condiciones adecuadas. La hibridación sonda-diana generalmente se detecta y cuantifica mediante detección basada en fluorescencia de dianas marcadas con fluoróforos para determinar la abundancia relativa de las secuencias de ácido nucleico diana . Microarray se ha utilizado para decodificar con éxito bibliotecas codificadas por ADN de ESAC y bibliotecas codificadas por PNA. Los oligonucleótidos codificantes que representan los compuestos químicos individuales en la biblioteca, se manchan y se unen químicamente en los portaobjetos de microarrays , utilizando un robot BioChip Arrayer. Posteriormente, las etiquetas de oligonucleótidos de los compuestos de unión aislados de la selección se amplifican por PCR usando un cebador fluorescente y se hibridan en el portaobjetos de microarrays de ADN . Posteriormente, los microarrays se analizan mediante un escaneo láser y se detectan y cuantifican las intensidades de los puntos. El enriquecimiento de los compuestos de unión preferencial se revela comparando la intensidad de los puntos del portaobjetos de microarrays de ADN antes y después de la selección.
Decodificación mediante secuenciación de alto rendimiento
De acuerdo con la complejidad de la biblioteca química codificada por ADN (típicamente entre 10 3 y 10 6 miembros), es poco probable que una decodificación convencional basada en secuenciación de Sanger sea utilizable en la práctica, debido tanto al alto costo por base para la secuenciación como al tedioso procedimiento involucrado. Las tecnologías de secuenciación de alto rendimiento explotaron estrategias que paralelizan el proceso de secuenciación desplazando el uso de electroforesis capilar y produciendo miles o millones de secuencias a la vez. En 2008 se describió la primera implementación de una técnica de secuenciación de alto rendimiento desarrollada originalmente para la secuenciación del genoma (es decir, " tecnología 454 ") para la decodificación rápida y eficiente de una biblioteca química codificada por ADN que comprende 4000 compuestos. Este estudio condujo a la identificación de nuevos compuestos químicos con constantes de disociación submicromolar hacia la estreptavidina y definitivamente demostró la viabilidad de construir, realizar selecciones y decodificar bibliotecas codificadas por ADN que contienen millones de compuestos químicos.
Ver también
- Descubrimiento de medicamento
- Proyección de alto impacto
- Química combinatoria
- secuencia ADN
- Visualización de fagos

